Топологически ассоциированный домен

Топологически ассоциированный домен ( TAD) — это самовзаимодействующая геномная область, что означает, что последовательности ДНК внутри TAD физически взаимодействуют друг с другом чаще, чем с последовательностями вне TAD. [1] Средний размер топологически ассоциированного домена (TAD) составляет 1000 кб у людей, 880 кб у мышей и 140 кб у плодовых мушек. [2] [3] Границы по обе стороны этих доменов сохраняются между различными типами клеток млекопитающих и даже между видами [2] и сильно обогащены фактором связывания CCCTC (CTCF) и когезином . [1] Кроме того, некоторые типы генов (такие как гены транспортной РНК и гены домашнего хозяйства ) появляются вблизи границ TAD чаще, чем можно было бы ожидать случайно. [4] [5]
Функции TAD до конца не изучены и все еще являются предметом дискуссий. Большинство исследований указывают на то, что TAD регулируют экспрессию генов , ограничивая взаимодействие энхансера и промоутера для каждого TAD; [6] однако недавнее исследование разделяет организацию TAD и экспрессию генов. [7] Обнаружено, что нарушение границ TAD связано с широким спектром заболеваний, таких как рак , [8] [9] [10] различными пороками развития конечностей, такими как синполидактилия , синдром Кука и F-синдром, [11] и рядом заболеваний головного мозга, таких как гипоплазия мозолистого тела и демиелинизирующая лейкодистрофия взрослого возраста. [11] Кроме того, исследования показали, что взаимодействия между промоторами и энхансерами, охватывающие один или несколько TAD, имеют основополагающее значение для точной динамики экспрессии генов. [12] Геномные элементы, лежащие в основе этих взаимодействий, называются дистальными элементами прикрепления (DTE), и было показано, что эти элементы важны для точной активации генов Hox в раннем эмбриогенезе D. melanogaster . [12]
Механизмы, лежащие в основе формирования TAD, также сложны и еще не полностью выяснены, хотя ряд белковых комплексов и элементов ДНК связаны с границами TAD. Однако модель наручников и модель экструзии петли описывают формирование TAD с помощью белков CTCF и когезина. [13] Кроме того, было высказано предположение, что жесткость границ TAD сама по себе может вызывать изоляцию домена и формирование TAD. [13]
Открытие и разнообразие
TAD определяются как регионы, последовательности ДНК которых преимущественно контактируют друг с другом. Они были обнаружены в 2012 году с использованием методов захвата конформации хромосом, включая Hi-C . [4] [14] [5] Было показано, что они присутствуют во многих видах, [15] включая плодовых мушек ( Drosophila ), [16] мышей , [4] растений, грибов и человека [5] геномы. У бактерий они называются доменами хромосомного взаимодействия (CID). [15]
Аналитические инструменты и базы данных
Местоположение TAD определяется путем применения алгоритма к данным Hi-C. Например, TAD часто называют в соответствии с так называемым «индексом направленности». [5] Индекс направленности рассчитывается для отдельных 40-килобайтных бинов путем сбора считываний, которые попадают в бин, и наблюдения за тем, отображаются ли их парные считывания вверх или вниз по течению от бина (пары считываний должны охватывать не более 2 Мб). Положительное значение указывает на то, что больше пар считываний лежит вниз по течению, чем вверх по течению, а отрицательное значение указывает на обратное. Математически индекс направленности представляет собой статистику хи-квадрат со знаком.
Разработка специализированных геномных браузеров и инструментов визуализации [17], таких как Juicebox, [18] HiGlass [19] /HiPiler, [20] 3D Genome Browser, [21] 3DIV, [22] 3D-GNOME, [23] и TADKB [24], позволила нам визуализировать TAD-организацию интересующих областей в различных типах клеток.
Механизмы формирования
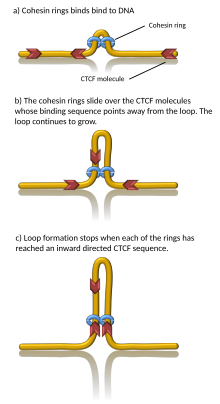
Известно, что ряд белков связаны с образованием TAD, включая белок CTCF и белковый комплекс когезин . [1] Также неизвестно, какие компоненты требуются на границах TAD; однако в клетках млекопитающих было показано, что эти пограничные области имеют сравнительно высокие уровни связывания CTCF. Кроме того, некоторые типы генов (такие как гены транспортной РНК и гены домашнего хозяйства ) появляются вблизи границ TAD чаще, чем можно было бы ожидать случайно. [4] [5]
Компьютерное моделирование показало, что выдавливание хроматиновых петель, управляемое моторами когезина, может генерировать TAD. [25] [26] В модели выдавливания петель когезин связывает хроматин, втягивает его и выдавливает хроматин, постепенно формируя петлю. Хроматин по обе стороны комплекса когезина выдавливается до тех пор, пока когезин не встретит связанный с хроматином белок CTCF, обычно расположенный на границе TAD. Таким образом, границы TAD могут быть объединены в качестве якорей хроматиновой петли. [27] Действительно, in vitro было замечено, что когезин процессивно выдавливает петли ДНК в зависимости от АТФ [28] [29] [30] и останавливается на CTCF. [31] [32] Однако некоторые данные in vitro указывают на то, что наблюдаемые петли могут быть артефактами. [33] [34] Важно отметить, что поскольку когезины могут динамически отсоединяться от хроматина, эта модель предполагает, что TAD (и связанные с ними петли хроматина) являются динамическими, временными структурами, [25] что согласуется с наблюдениями in vivo. [35] [36] [37] [38]
Были предложены и другие механизмы формирования TAD. Например, некоторые симуляции предполагают, что суперспирализация, генерируемая транскрипцией, может перемещать когезин к границам TAD [39] [40] или что пассивно диффундирующие «скользящие связи» когезина [41] [42] могут генерировать TAD.
Характеристики
Сохранение
Сообщалось, что TAD относительно постоянны между различными типами клеток (например, в стволовых клетках и клетках крови), а в отдельных случаях даже между видами. [5] [43] [44] [45] Сравнительный анализ TAD между Drosophila melanogaster и Drosophila subobscura , со временем расхождения приблизительно 49 миллионов лет, выявил сохранение в диапазоне 30-40%. [46]
Отношения с промоутерами и усилителями контактов
Большинство наблюдаемых взаимодействий между промоторами и энхансерами не пересекают границы TAD. Удаление границы TAD (например, с помощью CRISPR для удаления соответствующей области генома) может привести к образованию новых контактов промотор-энхансер. Это может повлиять на экспрессию генов поблизости — было показано, что такая неправильная регуляция вызывает пороки развития конечностей (например, полидактилию ) у людей и мышей. [44]
Компьютерное моделирование показало, что вызванная транскрипцией суперспирализация хроматиновых волокон может объяснить, как формируются TAD и как они могут обеспечивать очень эффективное взаимодействие между энхансерами и их родственными промоторами, расположенными в том же TAD. [39]
Связь с другими структурными особенностями генома
Было показано, что домены синхронизации репликации связаны с TAD, поскольку их граница локализуется совместно с границами TAD, которые расположены по обе стороны компартментов. [47] Изолированные окрестности , петли ДНК, образованные областями, связанными с CTCF/когезином, предположительно функционально лежат в основе TAD. [48]
Было показано, что точки разрыва перестройки генома обогащены на границах TAD у D. melanogaster . [49]
Роль в заболевании
Нарушение границ TAD может повлиять на экспрессию близлежащих генов, и это может вызвать заболевание. [50]
Например, сообщалось, что структурные варианты генома, которые нарушают границы TAD, вызывают нарушения развития, такие как пороки развития конечностей у человека. [51] [52] [53] Кроме того, несколько исследований предоставили доказательства того, что нарушение или перестройка границ TAD может обеспечить преимущества роста для некоторых видов рака, таких как острый лимфобластный лейкоз Т-клеток (T-ALL), [54] глиомы [55] и рак легких. [56]
Домены, ассоциированные с пластинкой

Домены, ассоциированные с пластинкой (LAD), представляют собой части хроматина, которые активно взаимодействуют с пластинкой, сетевидной структурой на внутренней мембране ядра . [57] LAD в основном состоят из транскрипционно молчащего хроматина, обогащенного триметилированным Lys27 на гистоне H3 (т. е. H3K27me3 ); что является распространенной посттрансляционной гистоновой модификацией гетерохроматина . [58] LAD имеют на своей периферии сайты связывания CTCF. [57]
Смотрите также
Ссылки
- ^ abc Pombo A, Dillon N (апрель 2015 г.). «Трехмерная архитектура генома: игроки и механизмы». Nature Reviews. Молекулярная клеточная биология . 16 (4): 245–257 . doi :10.1038/nrm3965. PMID 25757416. S2CID 6713103.
- ^ ab Yu M, Ren B (октябрь 2017 г.). «Трехмерная организация геномов млекопитающих». Annual Review of Cell and Developmental Biology . 33 : 265–289 . doi :10.1146/annurev-cellbio-100616-060531. PMC 5837811. PMID 28783961 .
- ^ Смирнов, Дмитрий Н.; Кононкова, Анна Д.; Тойбер, Дебра; Гельфанд, Михаил С.; Храмеева, Екатерина Е. (2024-01-29), optimizeTAD: аннотация топологически ассоциированных доменов на основе обогащения хроматиновых меток, doi :10.1101/2023.03.06.531254 , получено 2024-10-09
- ^ abcd Nora EP, Lajoie BR, Schulz EG, Giorgetti L, Okamoto I, Servant N и др. (апрель 2012 г.). «Пространственное разделение регуляторного ландшафта центра инактивации X». Nature . 485 (7398): 381– 385. Bibcode :2012Natur.485..381N. doi :10.1038/nature11049. PMC 3555144 . PMID 22495304.
- ^ abcdef Dixon JR, Selvaraj S, Yue F, Kim A, Li Y, Shen Y и др. (апрель 2012 г.). «Топологические домены в геномах млекопитающих, выявленные с помощью анализа взаимодействий хроматина». Nature . 485 (7398): 376– 380. Bibcode :2012Natur.485..376D. doi :10.1038/nature11082. PMC 3356448 . PMID 22495300.
- ^ Krijger PH, de Laat W (декабрь 2016 г.). «Регулирование экспрессии генов, связанных с заболеваниями, в трехмерном геноме». Nature Reviews. Molecular Cell Biology . 17 (12): 771– 782. doi :10.1038/nrm.2016.138. PMID 27826147. S2CID 11484886.
- ^ Ghavi-Helm Y; Jankowski A; Meiers S; Viales RR; Korbel JO ; Furlong EE (август 2019 г.). «Сильно перестроенные хромосомы обнаруживают разобщение между топологией генома и экспрессией генов». Nature Genetics . 51 (8): 1272– 1282. doi :10.1038/s41588-019-0462-3. PMC 7116017 . PMID 31308546.
- ^ Corces MR, Corces VG (февраль 2016 г.). «Трехмерный геном рака». Current Opinion in Genetics & Development . 36 : 1– 7. doi : 10.1016 /j.gde.2016.01.002. PMC 4880523. PMID 26855137.
- ^ Valton AL; Dekker J (февраль 2016 г.). «Нарушение TAD как онкогенный драйвер». Current Opinion in Genetics & Development . 36 : 34–40 . doi : 10.1016 /j.gde.2016.03.008. PMC 4880504. PMID 27111891.
- ^ Achinger-Kawecka J, Clark SJ (январь 2017 г.). «Нарушение трехмерной схемы генома рака». Epigenomics . 9 (1): 47–55 . doi : 10.2217/epi-2016-0111 . PMID 27936932.
- ^ ab Spielmann M, Lupiáñez DG, Mundlos S (июль 2018 г.). «Структурные вариации в трехмерном геноме». Nature Reviews. Genetics . 19 (7): 453– 467. doi : 10.1038/s41576-018-0007-0. hdl : 21.11116/0000-0003-610A-5 . PMID 29692413. S2CID 22325904.
- ^ ab Батут, Филипп Дж.; Бинг, Синь Ян; Сиско, Захари; Раймундо, Жуан; Лево, Михал; Левин, Майкл С. (2022-02-04). «Организация генома контролирует транскрипционную динамику во время развития». Science . 375 (6580): 566– 570. Bibcode :2022Sci...375..566B. doi :10.1126/science.abi7178. ISSN 0036-8075. PMC 10368186 . PMID 35113722.
- ^ ab Dixon JR, Gorkin DU, Ren B (июнь 2016 г.). «Домены хроматина: единица организации хромосомы». Molecular Cell . 62 (5): 668– 680. doi :10.1016/j.molcel.2016.05.018. PMC 5371509. PMID 27259200 .
- ^ de Laat W, Duboule D (октябрь 2013 г.). «Топология усилителей развития млекопитающих и их регуляторные ландшафты». Nature . 502 (7472): 499– 506. Bibcode :2013Natur.502..499D. doi :10.1038/nature12753. PMID 24153303. S2CID 4468533.
- ^ ab Szabo Q, Bantignies F, Cavalli G (апрель 2019 г.). "Принципы сворачивания генома в топологически ассоциированные домены". Science Advances . 5 (4): eaaw1668. Bibcode :2019SciA....5.1668S. doi :10.1126/sciadv.aaw1668. PMC 6457944 . PMID 30989119.
- ^ Sexton T, Yaffe E, Kenigsberg E, Bantignies F, Leblanc B, Hoichman M и др. (февраль 2012 г.). «Трехмерная укладка и принципы функциональной организации генома дрозофилы». Cell . 148 (3): 458– 472. doi : 10.1016/j.cell.2012.01.010 . PMID 22265598.
- ^ Ing-Simmons E, Vaquerizas JM (сентябрь 2019 г.). «Визуализация трехмерной организации генома в двух измерениях». Development . 146 (19): 99– 101. doi : 10.1242/dev.177162 . PMID 31558569.
- ^ Дюран NC; Робинсон JT; Шамим MS; Мачоль I; Месиров JP; Ландер ES; Эйден EL (июль 2016 г.). «Juicebox предоставляет систему визуализации для карт контактов Hi-C с неограниченным увеличением». Cell Systems . 3 (1): 99– 101. doi :10.1016/j.cels.2015.07.012. PMC 5596920 . PMID 27467250.
- ^ Kerpedjiev P, Abdennur N, Lekschas F, McCallum C, Dinkla K, Strobelt H и др. (август 2018 г.). "HiGlass: веб-визуальное исследование и анализ карт взаимодействия генома". Genome Biology . 19 (1): 125. doi : 10.1186/s13059-018-1486-1 . PMC 6109259 . PMID 30143029.
- ^ Lekschas F, Bach B, Kerpedjiev P, Gehlenborg N, Pfister H (январь 2018 г.). «HiPiler: визуальное исследование больших матриц взаимодействия генома с интерактивными малыми множественными». Труды IEEE по визуализации и компьютерной графике . 24 (1): 522– 531. doi : 10.1109/TVCG.2017.2745978. PMC 6038708. PMID 28866592 .
- ^ Wang Y, Song F, Zhang B, Zhang L, Xu J, Kuang D и др. (октябрь 2018 г.). «3D Genome Browser: веб-браузер для визуализации трехмерной организации генома и дальнодействующих взаимодействий хроматина». Genome Biology . 19 (1): 151. doi : 10.1186/s13059-018-1519-9 . PMC 6172833 . PMID 30286773.
- ^ Yang D, Jang I, Choi J, Kim MS, Lee AJ, Kim H и др. (январь 2018 г.). «3DIV: средство просмотра и база данных трехмерного взаимодействия генома». Nucleic Acids Research . 46 (D1): D52 – D57 . doi :10.1093/nar/gkx1017. PMC 5753379 . PMID 29106613.
- ^ Szalaj P, Michalski PJ, Wróblewski P, Tang Z, Kadlof M, Mazzocco G и др. (Июль 2016 г.). «3D-GNOME: интегрированный веб-сервис для структурного моделирования трехмерного генома». Nucleic Acids Research . 44 (W1): W288 – W293 . doi :10.1093/nar/gkw437. PMC 4987952 . PMID 27185892.
- ^ Лю, Т., Портер, Дж., Чжао, К. и др. TADKB: Классификация семейств и база знаний топологически ассоциированных доменов. BMC Genomics 20, 217 (2019). https://doi.org/10.1186/s12864-019-5551-2
- ^ аб Фуденберг Г., Имакаев М., Лу С., Голобородько А., Абденнур Н., Мирный Л.А. (май 2016 г.). «Формирование хромосомных доменов путем экструзии петель». Отчеты по ячейкам . 15 (9): 2038–2049 . doi :10.1016/j.celrep.2016.04.085. ПМЦ 4889513 . ПМИД 27210764.
- ^ Sanborn AL; Rao SS; Huang SC; Durand NC; Huntley MH; Jewett AI; et al. (Ноябрь 2015 г.). «Экструзия хроматина объясняет ключевые особенности формирования петель и доменов в геномах дикого типа и сконструированных геномах». Труды Национальной академии наук Соединенных Штатов Америки . 112 (47): E6456 – E6465 . Bibcode : 2015PNAS..112E6456S. doi : 10.1073/pnas.1518552112 . PMC 4664323. PMID 26499245 .
- ^ Яцкевич С.; Родс Дж.; Нейсмит К. (декабрь 2019 г.). «Организация хромосомной ДНК комплексами SMC». Annual Review of Genetics . 53 (1): 445–482 . doi : 10.1146/annurev-genet-112618-043633 . PMID 31577909. S2CID 203653572.
- ^ Golfier S, Quail T, Kimura H, Brugués J (май 2020 г.). Dekker, Struhl K, Mirny LA, Musacchio A, Marko JF (ред.). «Когезин и конденсин выдавливают петли ДНК в зависимости от клеточного цикла». eLife . 9 : e53885. doi : 10.7554/eLife.53885 . PMC 7316503 . PMID 32396063.
- ^ Davidson IF, Bauer B, Goetz D, Tang W, Wutz G, Peters JM (декабрь 2019 г.). «Экструзия петли ДНК человеческим когезином». Science . 366 (6471): 1338– 1345. Bibcode :2019Sci...366.1338D. doi : 10.1126/science.aaz3418 . PMID 31753851. S2CID 208228309.
- ^ Kim Y, Shi Z, Zhang H, Finkelstein IJ, Yu H (декабрь 2019 г.). «Человеческий когезин уплотняет ДНК путем экструзии петель». Science . 366 (6471): 1345– 1349. Bibcode :2019Sci...366.1345K. doi :10.1126/science.aaz4475. PMC 7387118 . PMID 31780627.
- ^ Дэвидсон ИФ, Барт Р, Зачек М, ван дер Торре Дж, Танг В, Нагасака К и др. (2022-09-09). «CTCF — это зависящий от натяжения ДНК барьер для когезин-опосредованной экструзии петли ДНК». bioRxiv 10.1101/2022.09.08.507093 .
- ^ Чжан Х, Ши З, Баниган Э.Дж., Ким И., Ю Х, Бай Х, Финкельштейн И.Дж. (2022-10-07). "CTCF и R-петли являются границами опосредованного когезином петлеобразования ДНК". bioRxiv 10.1101/2022.09.15.508177 .
- ^ Man, Zhou (сентябрь 2022 г.). «Скольжение ДНК и образование петель комплексом E. coli SMC: MukBEF». Biochemistry and Biophysics Reports . 31 : 101297. doi : 10.1016/j.bbrep.2022.101297. PMC 9234588. PMID 35770038 .
- ^ Рю Дж.К., Бушу С., Лю Х.В., Ким Э., Минамино М., де Гроот Р., Катан А.Дж., Бонато А., Марендуццо Д., Микиелетто Д., Ульманн Ф. (февраль 2021 г.). «Мостиковое разделение фаз, индуцированное белковыми комплексами когезина SMC». Достижения науки . 7 (7): eabe5905. Бибкод : 2021SciA....7.5905R. doi : 10.1126/sciadv.abe5905. ПМЦ 7875533 . ПМИД 33568486.
- ^ Габриэле М, Брандао ХБ, Гроссе-Хольц С, Джа А, Дейли ГМ, Каттоглио К и др. (апрель 2022 г.). «Динамика CTCF- и когезин-опосредованного хроматинового петельчатого образования, выявленная с помощью визуализации живых клеток». Science . 376 (6592): 496– 501. Bibcode :2022Sci...376..496G. doi :10.1126/science.abn6583. PMC 9069445 . PMID 35420890.
- ^ Беквит КС, Эдегард-Фоугнер О, Мореро НР, Бартон К, Шудер Ф, Танг В и др. (2022-05-02). «Визуализация экструзии петли с помощью наномасштабной трассировки ДНК в отдельных клетках человека». bioRxiv 10.1101/2021.04.12.439407 .
- ^ Mach P, Kos PI, Zhan Y, Cramard J, Gaudin S, Tünnermann J и др. (2022-03-03). «Визуализация живых клеток и физическое моделирование выявляют контроль динамики сворачивания хромосом когезином и CTCF». bioRxiv 10.1101/2022.03.03.482826 .
- ^ Флямер И.М., Гасслер Дж., Имакаев М., Брандао Х.Б., Ульянов С.В., Абденнур Н. и др. (апрель 2017 г.). «Одноядерный Hi-C обнаруживает уникальную реорганизацию хроматина при переходе от ооцита к зиготе». Природа . 544 (7648): 110–114 . Бибкод : 2017Natur.544..110F. дои : 10.1038/nature21711. ПМЦ 5639698 . ПМИД 28355183.
- ^ ab Racko D, Benedetti F , Dorier J, Stasiak A (январь 2019). «Являются ли TAD суперспиральными?». Nucleic Acids Research . 47 (2): 521– 532. doi :10.1093/nar/gky1091. PMC 6344874. PMID 30395328.
- ^ Рако Д., Бенедетти Ф., Дорье Дж., Стасиак А. (февраль 2018 г.). «Транскрипционно-индуцированная суперспирализация как движущая сила экструзии петель хроматина во время образования TAD в интерфазных хромосомах». Nucleic Acids Research . 46 (4): 1648– 1660. doi :10.1093/nar/gkx1123. PMC 5829651. PMID 29140466 .
- ^ Brackley CA, Johnson J, Michieletto D, Morozov AN, Nicodemi M, Cook PR, Marenduzzo D (сентябрь 2017 г.). "Неравновесное зацикливание хромосом с помощью молекулярных скользящих связей". Physical Review Letters . 119 (13): 138101. arXiv : 1612.07256 . Bibcode :2017PhRvL.119m8101B. doi :10.1103/PhysRevLett.119.138101. PMID 29341686. S2CID 14706723.
- ^ Yamamoto T, Schiessel H (сентябрь 2017 г.). «Осмотический механизм процесса экструзии петли». Physical Review E. 96 ( 3– 1 ): 030402. Bibcode : 2017PhRvE..96c0402Y. doi : 10.1103/PhysRevE.96.030402. hdl : 1887/58394 . PMID 29346962.
- ^ Vietri Rudan M, Barrington C, Henderson S, Ernst C, Odom DT, Tanay A, Hadjur S (март 2015 г.). «Сравнительный Hi-C показывает, что CTCF лежит в основе эволюции архитектуры хромосомных доменов». Cell Reports . 10 (8): 1297– 1309. doi :10.1016/j.celrep.2015.02.004. PMC 4542312 . PMID 25732821.
- ^ ab Jost D, Vaillant C, Meister P (февраль 2017 г.). «Связь модификаций 1D и организации ядра 3D: данные, модели и функции». Current Opinion in Cell Biology . 44 : 20–27 . doi :10.1016/j.ceb.2016.12.001. PMID 28040646.
- ^ Yang Y, Zhang Y, Ren B, Dixon JR, Ma J (июнь 2019 г.). «Сравнение трехмерной организации генома у нескольких видов с использованием Phylo-HMRF». Cell Systems . 8 (6): 494–505.e14. doi :10.1016/j.cels.2019.05.011. PMC 6706282 . PMID 31229558.
- ^ Ляо, И; Чжан, Синьвэнь; Чакраборти, Махул; Эмерсон, Дж. Дж. (2021-03-01). «Топологически ассоциированные домены и их роль в эволюции структуры и функции генома у дрозофилы». Genome Research . 31 (3): 397– 410. doi :10.1101/gr.266130.120. ISSN 1088-9051. PMC 7919452 . PMID 33563719.
- ^ Marchal C, Sima J, Gilbert DM (декабрь 2019 г.). «Контроль времени репликации ДНК в трехмерном геноме». Nature Reviews. Molecular Cell Biology . 20 (12): 721– 737. doi :10.1038/s41580-019-0162-y. PMC 11567694. PMID 31477886. S2CID 201714312 .
- ^ Ji X, Dadon DB, Powell BE, Fan ZP, Borges-Rivera D, Shachar S и др. (февраль 2016 г.). «3D-регуляторный ландшафт хромосом плюрипотентных клеток человека». Cell Stem Cell . 18 (2): 262– 275. doi :10.1016/j.stem.2015.11.007. PMC 4848748 . PMID 26686465.
- ^ Ляо, И; Чжан, Синьвэнь; Чакраборти, Махул; Эмерсон, Дж. Дж. (2021-03-01). «Топологически ассоциированные домены и их роль в эволюции структуры и функции генома у дрозофилы». Genome Research . 31 (3): 397– 410. doi :10.1101/gr.266130.120. ISSN 1088-9051. PMC 7919452 . PMID 33563719.
- ^ Lupiáñez DG, Spielmann M, Mundlos S (апрель 2016 г.). «Нарушение TAD: как изменения хроматиновых доменов приводят к болезням». Trends in Genetics . 32 (4): 225–237 . doi :10.1016/j.tig.2016.01.003. hdl : 11858/00-001M-0000-002E-1D1D-D . PMID 26862051.
- ^ Lupiáñez DG, Kraft K, Heinrich V, Krawitz P, Brancati F, Klopocki E и др. (май 2015 г.). «Нарушения топологических доменов хроматина вызывают патогенную перестройку взаимодействий генов-энхансеров». Cell . 161 (5): 1012– 1025. doi :10.1016/j.cell.2015.04.004. PMC 4791538 . PMID 25959774.
- ^ Angier N (2017-01-09). «Общий дефект семьи проливает свет на геном человека». The New York Times .
- ^ Franke M, Ibrahim DM, Андрей G, Schwarzer W, Heinrich V, Schöpflin R и др. (октябрь 2016 г.). «Формирование новых доменов хроматина определяет патогенность геномных дупликаций». Nature . 538 (7624): 265– 269. Bibcode :2016Natur.538..265F. doi :10.1038/nature19800. hdl : 11858/00-001M-0000-002C-010A-3 . PMID 27706140. S2CID 4463482.
- ^ Hnisz D, Weintraub AS, Day DS, Valton AL, Bak RO, Li CH и др. (март 2016 г.). «Активация протоонкогенов путем нарушения хромосомных окрестностей». Science . 351 (6280): 1454– 1458. Bibcode :2016Sci...351.1454H. doi :10.1126/science.aad9024. PMC 4884612 . PMID 26940867.
- ^ Флавахан В.А., Драйер Ю., Лиау Б.Б., Гиллеспи С.М., Вентайхер А.С., Стеммер-Рахамимов А.О. и др. (январь 2016 г.). «Инсуляторная дисфункция и активация онкогенов в глиомах с мутацией IDH». Природа . 529 (7584): 110–114 . Бибкод : 2016Natur.529..110F. дои : 10.1038/nature16490. ПМЦ 4831574 . ПМИД 26700815.
- ^ Weischenfeldt J, Dubash T, Drainas AP, Mardin BR, Chen Y, Stütz AM и др. (январь 2017 г.). «Анализ изменений числа соматических копий в целом по раку указывает на связь IRS4 и IGF2 с захватом энхансера». Nature Genetics . 49 (1): 65–74 . doi :10.1038/ng.3722. PMC 5791882 . PMID 27869826.
- ^ ab Gonzalez-Sandoval A; Gasser SM (август 2016 г.). «О TAD и LAD: пространственный контроль над экспрессией генов». Trends in Genetics . 32 (8): 485– 495. doi :10.1016/j.tig.2016.05.004. PMID 27312344.
- ^ Li M, Liu GH, Izpisua Belmonte JC (июль 2012 г.). «Навигация по эпигенетическому ландшафту плюрипотентных стволовых клеток». Nature Reviews. Молекулярная клеточная биология . 13 (8): 524– 535. doi :10.1038/nrm3393. PMID 22820889. S2CID 22524502.
Внешние ссылки
- Соковыжималка
- HiGlass
- HiPiler
- Браузер 3D-генома
- 3ДИВ
- 3D-ГНОМ
- ТАДКБ