Комбинированный бисульфитный рестрикционный анализ
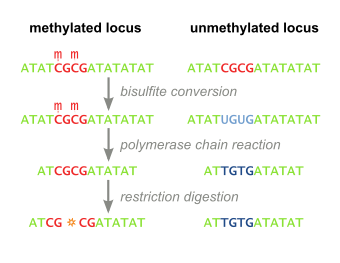
Комбинированный бисульфитный рестрикционный анализ (или COBRA ) — это метод молекулярной биологии, который позволяет проводить чувствительную количественную оценку уровней метилирования ДНК в определенном геномном локусе последовательности ДНК в небольшом образце геномной ДНК. [1] Метод представляет собой разновидность бисульфитного секвенирования и сочетает полимеразную цепную реакцию на основе бисульфитной конверсии с рестрикционным расщеплением . Первоначально разработанный для надежной обработки мельчайших количеств геномной ДНК из микропрепарированных образцов тканей, залитых парафином , метод с тех пор нашел широкое применение в исследованиях рака и эпигенетических исследованиях. [2]
Процедура
Обработка бисульфитом
Геномная ДНК интереса обрабатывается бисульфитом натрия , который вносит различия в последовательности, зависящие от метилирования. Во время обработки бисульфитом натрия неметилированные остатки цитозина преобразуются в урацил , тогда как метилированные остатки цитозина остаются неизменными.

ПЦР-амплификация

Затем обработанная бисульфитом ДНК амплифицируется методом ПЦР, что приводит к образованию остатков цитозина в изначально метилированных позициях и остатков тимина в изначально неметилированной позиции (которые были преобразованы в урацил). Праймеры, используемые на этом этапе, не содержат сайтов CpG (общая цель метилирования цитозина), поэтому процесс амплификации не различает шаблоны на основе статуса метилирования. Продукты ПЦР очищаются для обеспечения полного переваривания на следующем этапе.
Дайджест ограничений
Вышеуказанные шаги приводят к зависимому от метилирования сохранению или потере сайтов рестриктаз , содержащих CpG , таких как сайты для TaqI (TCGA) и BstUI (CGCG), в зависимости от того, был ли остаток цитозина изначально метилирован или нет, соответственно. Из-за независимой от метилирования амплификации на вышеуказанном шаге полученные продукты ПЦР будут представлять собой смешанную популяцию фрагментов, которые потеряли или сохранили сайты рестриктаз, содержащие CpG, чьи соответствующие проценты будут напрямую коррелировать с исходным уровнем метилирования ДНК в образце ДНК.
Затем продукты ПЦР обрабатываются рестриктазой ( например, BstUI), которая расщепляет только изначально метилированные сайты (CGCG), оставляя изначально неметилированные сайты (TGTG). Чтобы гарантировать, что все сайты CpG сохраняются из-за изначального метилирования, а не являются остатками неполного бисульфитного превращения, проводится контрольное расщепление с помощью таких ферментов, как Hsp92II, который распознает последовательность CATG, ни один из которых не должен оставаться после бисульфитного превращения (за редким исключением не-CpG метилирования), и, таким образом, расщепления не должно происходить, если бисульфитное превращение было полным.
Количественная оценка
Затем расщепленные фрагменты разделяются электрофорезом в полиакриламидном геле с ожидаемым появлением полос, соответствующих одному большому нерасщепленному фрагменту, и нескольких меньших полос, соответствующих расщепленным фрагментам. Количественное количество ДНК в этих полосах можно определить с помощью такого устройства, как фосфоимиджер , после чего процент метилирования исходного образца можно рассчитать по формуле:
Использование и применение
COBRA широко используется во многих исследовательских приложениях, таких как скрининг изменений метилирования ДНК в промоторах генов в исследованиях рака, [3] [4] обнаружение измененных паттернов метилирования в импринтированных генах , [5] и характеристика паттернов метилирования в геноме во время развития млекопитающих. [6] [7]
В медицине COBRA использовался как инструмент для диагностики заболеваний человека, связанных с аберрантным метилированием ДНК. Исследователи использовали COBRA в сочетании с денатурирующей высокоэффективной жидкостной хроматографией для диагностики генетического импринтингового расстройства синдрома Рассела-Сильвера , где гипометилирование импринтированного гена H19 является причиной расстройства у 50% пациентов. [8]
Сильные стороны
- Просто, быстро и недорого : в COBRA уровни метилирования ДНК легко и быстро измеряются без необходимости трудоемкого субклонирования и секвенирования , как при бисульфитном секвенировании. Анализ прост и может быть выполнен с использованием стандартных недорогих реагентов молекулярной биологии.
- Высокая совместимость : благодаря этапам ПЦР и очистки метод работает не только с очень малыми количествами геномной ДНК, но и с образцами, обработанными парафином, что может создавать проблемы в других протоколах количественной оценки метилирования ДНК, таких как Саузерн-блоттинг и расщепление рестрикционными ферментами, чувствительными к метилированию, с последующей ПЦР.
- Количественный : Это в отличие от ПЦР, специфичной для метилирования , которая является качественной. С помощью COBRA уровни метилирования ДНК могут быть напрямую количественно определены в заданном локусе, что дает больше информации за анализ.
- Масштабируемость для высокопроизводительной обработки образцов : с COBRA многие интересующие регионы могут обрабатываться параллельно в отдельных образцах, переваренных одним и тем же рестриктазой. Это отличается от анализа бисульфитного секвенирования, где каждый регион должен быть тщательно исследован путем секвенирования многих клонов на локус, что требует больше времени.
- Несколько запросов на анализ : статус метилирования можно проверить на нескольких сайтах рестрикции, содержащих CpG, в одном анализе переваривания.
Слабые стороны
- Анализ ограничен использованием существующих сайтов рестрикции в интересующей области, и метилирование, которое не происходит в контексте определенного сайта рестрикции, не будет анализироваться.
- Неполное переваривание рестриктазами после ПЦР может запутать анализ: неполное переваривание будет предполагать отсутствие метилирования ДНК (если разрезание производится ферментом, чувствительным к метилированию, таким как HpaII). Также известно, что BstUI может резать в неконвертированных участках, что приводит к завышению уровня метилирования, поэтому часто необходимо использование HpaII. [9]
- В сложных образцах гетерогенность типа клеток может запутать анализ , поскольку ДНК не секвенируется, гетерогенность последовательностей из разных клеток в образце ( т. е. разных популяций клеток в опухоли), которые приобрели мутации в исследуемой области, такие как изменение динуклеотида CG на CA или CT, приведет к потере сайта рестрикции, что приведет к появлению явно метилированной области из-за отсутствия переваривания. Это исказит количественную оценку уровней метилирования ДНК в данном образце.
Альтернативы
В целом, COBRA часто сочетается с другими анализами метилирования ДНК и часто используется при первоначальном скрининге интересующих локусов. Если COBRA предполагает измененные паттерны метилирования, то можно применять более строгие, трудоемкие методы, такие как бисульфитное секвенирование или MeDIP . Также для обнаружения метилирования ДНК можно использовать секвенирование PacBio.
Ссылки
- ^ Сюн, Чжэнган; Лэрд, Питер В. (1997). «COBRA: чувствительный и количественный анализ метилирования ДНК». Nucleic Acids Research . 25 (12): 2532– 2534. doi :10.1093/nar/25.12.2532. PMC 146738. PMID 9171110 .
- ^ Лэрд, Питер В. (2003). «Сила и перспективы маркеров метилирования ДНК». Nature Reviews Cancer . 3 (апрель): 253–266 . doi :10.1038/nrc1045. PMID 12671664. S2CID 19574628.По данным Scopus , на эту статью по состоянию на 2–11 февраля ссылались 576 раз.
- ^ Шен, Ланлан и др. (2005). «MGMT Promoter Methylation and Field Defect in Sporadic Colorectal Cancer». Журнал Национального института рака . 97 (18): 1330–1338 . CiteSeerX 10.1.1.536.6096 . doi :10.1093/jnci/dji275. PMID 16174854.
- ^ Suter, Catherine, M.; et al. (2004). «Эпимутация зародышевой линии в MLH1 у лиц с множественными раковыми заболеваниями». Nature Genetics . 36 (5): 497– 501. doi : 10.1038/ng1342 . PMID 15064764.
{{cite journal}}: CS1 maint: несколько имен: список авторов ( ссылка ) - ^ Bliek, Jet; et al. (2009). "Гипометилирование в множественных матерински метилированных импринтированных регионах, включая локусы PLAGL1 и GNAS при синдроме Беквита-Видеманна, и характеристика паттернов метилирования ДНК в геноме во время развития млекопитающих" (PDF) . European Journal of Human Genetics . 17 (5): 611– 618. doi :10.1038/ejhg.2008.233. PMC 2986258 . PMID 19092779.
- ^ Wernig, Marius; et al. (2007). «In vitro перепрограммирование фибробластов в состояние, подобное плюрипотентным ES-клеткам». Nature . 448 (7151): 318– 324. Bibcode :2007Natur.448..318W. doi :10.1038/nature05944. PMID 17554336. S2CID 4377572.
- ^ Миккельсен, Тарьей С.; и др. (2008). «Раскрытие прямого перепрограммирования посредством интегративного геномного анализа». Nature . 454 (июль): 49–56 . Bibcode :2008Natur.454...49M. doi :10.1038/nature07056. PMC 2754827 . PMID 18509334.
- ^ Хаттори, М.; и др. (2009). «Диагностика синдрома Рассела-Сильвера с помощью комбинированного анализа рестрикции бисульфита и высокоэффективной жидкостной хроматографии с денатурацией». Генетическое тестирование и молекулярные биомаркеры . 13 (5): 623– 630. doi :10.1089/gtmb.2009.0018. PMID 19814617.
- ^ Фрага, Марио Ф.; Эстеллер, Манель (2002). «Метилирование ДНК: профиль методов и приложений». BioTechniques . 33 (3): 633– 649. doi : 10.2144/02333rv01 . PMID 12238773.
