Иммунопреципитация метилированной ДНК
Иммунопреципитация метилированной ДНК (MeDIP или mDIP) — это крупномасштабный ( хромосомный или геномный ) метод очистки в молекулярной биологии, который используется для обогащения метилированных последовательностей ДНК . Он заключается в выделении метилированных фрагментов ДНК с помощью антитела, полученного против 5-метилцитозина (5mC). Этот метод был впервые описан Вебером М. и др. [1] в 2005 году и помог проложить путь для жизнеспособных усилий по оценке на уровне метилома , поскольку очищенная фракция метилированной ДНК может быть введена в высокопроизводительные методы обнаружения ДНК, такие как ДНК-микрочипы высокого разрешения ( MeDIP-chip ) или секвенирование следующего поколения (MeDIP-seq). Тем не менее, понимание метилома остается рудиментарным; его изучение осложняется тем фактом, что, как и другие эпигенетические свойства, паттерны различаются от типа клеток к типу клеток.
Фон
Метилирование ДНК , относящееся к обратимому метилированию 5-го положения цитозина метилтрансферазами , является основной эпигенетической модификацией в многоклеточных организмах. [2] У млекопитающих эта модификация в основном происходит в сайтах CpG , которые, в свою очередь, имеют тенденцию группироваться в областях, называемых CpG-островками . [3] Существует небольшая часть CpG-островков, которые могут перекрываться или находиться в непосредственной близости от промоторных областей сайтов начала транскрипции. Модификация может также происходить в других сайтах, [4] но метилирование в любом из этих сайтов может подавлять экспрессию генов, либо препятствуя связыванию факторов транскрипции , либо изменяя структуру хроматина до репрессивного состояния. [5]
Исследования состояний болезни в значительной степени подпитывали усилия по пониманию роли метилирования ДНК. В настоящее время основной исследовательский интерес заключается в изучении состояний болезни, таких как рак, для выявления областей ДНК, которые претерпели обширные изменения метилирования. Гены, содержащиеся в этих областях, представляют функциональный интерес, поскольку они могут предложить механистическое объяснение основных генетических причин заболевания. Например, аномальный паттерн метилирования раковых клеток [6] [7] [8] изначально был показан как механизм, посредством которого гены, подобные супрессорам опухолей , подавляются [9] , хотя позже было замечено, что затронут гораздо более широкий диапазон типов генов. [10] [11] [12]
Другие технологии
Существует два подхода к анализу метилирования: технологии типирования и профилирования. Технологии типирования нацелены на небольшое количество локусов во многих образцах и включают использование таких методов, как ПЦР , рестриктазы и масс-спектрометрия . Технологии профилирования, такие как MeDIP, нацелены на оценку метилирования на уровне генома или метилома ; это включает геномное сканирование рестрикционных маркеров (RLGS) [13] и методы, основанные на бисульфитной конверсии , которые основаны на обработке ДНК бисульфитом для преобразования неметилированных остатков цитозина в урацил . [14] [15] [16] [17]
Ограничения других технологий
Другие методы картирования и профилирования метилома были эффективны, но не лишены своих ограничений, которые могут повлиять на разрешение, уровень пропускной способности или экспериментальные вариации. Например, RLGS ограничен количеством сайтов рестрикции в геноме, которые могут быть целями для рестриктазы; как правило, можно оценить максимум ~4100 ориентиров. [18] Методы, основанные на бисульфитном секвенировании , несмотря на возможное разрешение по одному нуклеотиду, имеют недостаток: преобразование неметилированного цитозина в урацил может быть нестабильным. [19] Кроме того, когда бисульфитное преобразование сочетается с ДНК-микрочипами для обнаружения сайтов, преобразованных бисульфитом, проблемой становится сниженная сложность последовательности ДНК. Микрочипы, способные всесторонне профилировать весь геном, становится трудно разрабатывать, поскольку доступно меньше уникальных зондов. [20]
Методы
В следующих разделах описывается метод MeDIP в сочетании с гибридизацией массивов высокого разрешения или высокопроизводительным секвенированием. Каждый метод обнаружения ДНК также кратко описывает постлабораторную обработку и анализ. Требуется различная постобработка необработанных данных в зависимости от технологии, используемой для идентификации метилированных последовательностей. Это аналогично данным, полученным с помощью ChIP-chip и ChIP-seq .
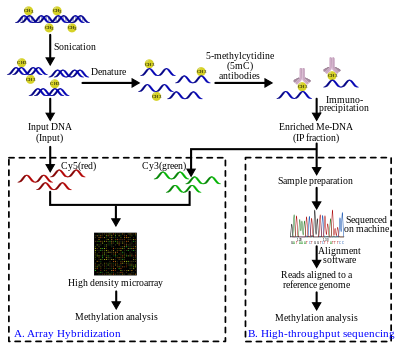
Иммунопреципитация метилированной ДНК (MeDIP)
Геномная ДНК извлекается ( экстракция ДНК ) из клеток и очищается. Затем очищенная ДНК подвергается ультразвуковой обработке для ее разрезания на случайные фрагменты. Этот процесс ультразвуковой обработки быстрый, простой и позволяет избежать смещений рестриктаз . Длина полученных фрагментов составляет от 300 до 1000 пар оснований (п. н.), хотя обычно они составляют от 400 до 600 п. н. [21] Короткая длина этих фрагментов важна для получения адекватного разрешения, повышения эффективности последующего этапа иммунопреципитации и снижения эффектов или смещений длины фрагмента. Кроме того, размер фрагмента влияет на связывание антитела 5-метилцитидина (5mC), поскольку для эффективного связывания антителу требуется больше, чем просто один 5mC. [22] Для дальнейшего улучшения сродства связывания антител фрагменты ДНК денатурируют для получения одноцепочечной ДНК. После денатурации ДНК инкубируют с моноклональными антителами 5mC. Затем применяется классический метод иммунопреципитации : магнитные шарики, конъюгированные с антимышиным IgG, используются для связывания анти-5mC антител, а несвязанная ДНК удаляется в супернатанте. Для очистки ДНК добавляется протеиназа К для расщепления антител и высвобождения ДНК, которую можно собрать и подготовить для обнаружения ДНК.
Более подробную информацию об экспериментальных этапах см. [1] [19] [23] [24]
MeDIP и гибридизация на основе массива (MeDIP-чип)
Часть входной ДНК, полученной после этапа обработки ультразвуком, маркируется цианином -5 (Cy5; красный) дезокси-цитозин-трифосфатом, в то время как метилированная ДНК, обогащенная после этапа иммунопреципитации, маркируется цианином -3 (Cy3; зеленый). Образцы маркированной ДНК когибридизуются на 2-канальном геномном микрочипе высокой плотности для зондирования на наличие и относительные количества. Целью этого сравнения является выявление последовательностей, которые показывают значительные различия в уровнях гибридизации, тем самым подтверждая, что интересующая последовательность обогащена. Идентификация последовательностей MeDIP на основе массива ограничена конструкцией массива. В результате разрешение ограничивается зондами в конструкции массива. Существуют дополнительные стандартные шаги, необходимые при обработке сигнала для исправления проблем гибридизации, таких как шум, как в случае с большинством технологий массивов.
Более подробную информацию см. в [23] [24] [25] .
MeDIP и высокопроизводительное секвенирование (MeDIP-seq)
Подход MeDIP-seq, т. е. соединение MeDIP с технологиями секвенирования с короткими прочтениями следующего поколения, такими как пиросеквенирование 454 или Illumina (Solexa), был впервые описан Дауном и др. в 2008 году. [20] Высокопроизводительное секвенирование метилированных фрагментов ДНК производит большое количество коротких прочтений (36-50 п.н. [26] или 400 п.н. [27] в зависимости от технологии). Короткие прочтения выравниваются с эталонным геномом с помощью программного обеспечения для выравнивания, такого как Mapping and Assembly with Quality (Maq), которое использует байесовский подход, а также базовые и картографические качества для моделирования вероятностей ошибок для выравниваний. [28] Затем прочтения можно расширить, чтобы представить фрагменты размером ~400–700 п.н. с этапа ультразвуковой обработки. Охват этих расширенных прочтений можно использовать для оценки уровня метилирования региона. Для визуализации данных можно также использовать геномный браузер, такой как Ensembl .
Валидацию подхода к оценке качества и точности данных можно выполнить с помощью количественной ПЦР . Это делается путем сравнения последовательности из образца MeDIP с неметилированной контрольной последовательностью. Затем образцы прогоняются на геле и сравниваются интенсивности полос. [19] Относительная интенсивность служит руководством для поиска обогащения. Результаты также можно сравнить с результатами MeDIP-чипа, чтобы помочь определить необходимое покрытие.
Анализ биоинформатики в нисходящем направлении

Оценки уровня метилирования ДНК могут быть искажены из-за различной плотности метилированных сайтов CpG по всему геному при наблюдении за данными, полученными с помощью MeDIP. Это может быть проблематичным для анализа регионов с низким содержанием CpG (более низкой плотности). Одной из причин этой проблемы с плотностью является ее влияние на эффективность иммунопреципитации. В своем исследовании Даун и др. [20] разработали инструмент для оценки абсолютных уровней метилирования из данных, полученных с помощью MeDIP, путем моделирования плотности метилированных сайтов CpG. Этот инструмент называется байесовским инструментом для анализа метилирования (Batman) . В исследовании сообщается об охвате ~90% всех сайтов CpG в промоторах, областях кодирования генов, островах и регуляторных элементах, где можно оценить уровни метилирования; это почти в 20 раз лучшее покрытие, чем у любых предыдущих методов.
Исследования с использованием MeDIP-seq или MeDIP-chip являются подходами, охватывающими весь геном, которые имеют общую цель получения функционального картирования метилома. После того, как области метилирования ДНК идентифицированы, можно применить ряд биоинформатических анализов для ответа на определенные биологические вопросы. Одним из очевидных шагов является исследование генов, содержащихся в этих областях, и изучение функциональной значимости их репрессии. Например, подавление генов-супрессоров опухолей при раке может быть отнесено к метилированию ДНК. [29] Выявляя мутационные события, приводящие к гиперметилированию и последующей репрессии известных генов-супрессоров опухолей, можно более конкретно охарактеризовать факторы, способствующие причине заболевания. В качестве альтернативы можно идентифицировать гены, которые, как известно, обычно метилированы, но в результате некоторого мутационного события больше не подавлены.
Кроме того, можно попытаться исследовать и определить, был ли затронут какой-либо эпигенетический регулятор, такой как ДНК-метилтрансфераза (DNMT); [21] в этих случаях обогащение может быть более ограниченным.
Было показано, что анализ набора генов (например, с использованием таких инструментов, как DAVID и GoSeq) является сильно предвзятым при применении к данным метилирования с высокой пропускной способностью (например, MeDIP-seq и MeDIP-ChIP); было высказано предположение, что это можно исправить с помощью перестановок меток образцов или с помощью статистической модели для контроля различий в количестве зондов CpG / сайтов CpG, нацеленных на каждый ген. [30]
Ограничения MeDIP
Ограничения, которые следует учитывать при использовании MeDIP, являются типичными экспериментальными факторами. Они включают качество и перекрестную реактивность антител 5mC, используемых в процедуре. Кроме того, методы обнаружения ДНК (т. е. гибридизация массивов и высокопроизводительное секвенирование) обычно включают в себя хорошо известные ограничения. В частности, для процедур на основе массивов, как упоминалось выше, анализируемые последовательности ограничены конкретной используемой конструкцией массива.
Наиболее типичные ограничения применимы к высокопроизводительному секвенированию следующего поколения. Проблема точности выравнивания для повторяющихся областей в геноме приведет к менее точному анализу метилирования в этих областях. Кроме того, как было упомянуто выше, короткие считывания (например, 36-50 п.н. из анализатора генома Illumina) представляют собой часть срезанного фрагмента при выравнивании с геномом; поэтому точный сайт метилирования может попасть в любое место в пределах окна, которое является функцией размера фрагмента. [19] В этом отношении бисульфитное секвенирование имеет гораздо более высокое разрешение (вплоть до одного сайта CpG; уровень одного нуклеотида). Однако этот уровень разрешения может не потребоваться для большинства приложений, поскольку было показано, что статус метилирования сайтов CpG в пределах < 1000 п.н. значительно коррелирует. [20]
Применение MeDIP
- Вебер и др. 2005 [1] с помощью MeDIP в сочетании с микрочипом определили, что неактивная Х-хромосома у женщин гиперметилирована на уровне всей хромосомы.
- Кешет и др. 2006 [31] провели исследование клеток рака толстой кишки и простаты с использованием MeDIP-чипа. Результатом стал полногеномный анализ генов, лежащих в гиперметилированных регионах, а также заключение о наличии поучительного механизма de novo метилирования в раковых клетках.
- Чжан и др. 2006 [24] получили метиломное картирование высокого разрешения в Arabidopsis с использованием MeDIP-чипа.
- Новак и др. 2006 [32] использовали подход MeDIP-chip для исследования рака молочной железы человека на предмет подавления, связанного с метилированием, и наблюдали инактивацию кластера генов HOXA
Смотрите также
Ссылки
- ^ abc Вебер М., Дэвис Дж. Дж., Виттиг Д. и др. (август 2005 г.). «Хромосомный и промоторно-специфический анализы выявляют участки дифференциального метилирования ДНК в нормальных и трансформированных клетках человека». Nat. Genet . 37 (8): 853–62 . doi :10.1038/ng1598. PMID 16007088. S2CID 14505320.
- ^ Bird A (январь 2002 г.). «Паттерны метилирования ДНК и эпигенетическая память». Genes Dev . 16 (1): 6–21 . doi : 10.1101/gad.947102 . PMID 11782440.
- ^ Гардинер-Гарден М, Фроммер М (июль 1987). «CpG-островки в геномах позвоночных». J. Mol. Biol . 196 (2): 261– 82. doi :10.1016/0022-2836(87)90689-9. PMID 3656447.
- ^ Clark SJ, Harrison J, Frommer M (май 1995). "CpNpG метилирование в клетках млекопитающих". Nat. Genet . 10 (1): 20– 7. doi :10.1038/ng0595-20. PMID 7647784. S2CID 8857760.
- ^ Jaenisch R, Bird A (март 2003 г.). «Эпигенетическая регуляция экспрессии генов: как геном интегрирует внутренние и внешние сигналы». Nat. Genet . 33 (Suppl): 245–54 . doi :10.1038/ng1089. PMID 12610534. S2CID 17270515.
- ^ Робертсон КД, Вольфе А.П. (октябрь 2000 г.). «Метилирование ДНК в здоровье и болезни». Nat. Rev. Genet . 1 (1): 11– 9. doi :10.1038/35049533. PMID 11262868. S2CID 1915808.
- ^ Baylin SB , Herman JG (2000). «Гиперметилирование ДНК в опухолеобразовании: эпигенетика присоединяется к генетике». Trends Genet . 16 (4): 268–274 . doi : 10.1016/S0168-9525(99)01971-X . PMID 10729832.
- ^ Jones PA, Laird PW (февраль 1999). «Эпигенетика рака достигает зрелости». Nat. Genet . 21 (2): 163– 7. doi :10.1038/5947. PMID 9988266. S2CID 10328397.
- ^ Jones PA, Baylin SB (июнь 2002 г.). «Фундаментальная роль эпигенетических событий в раке». Nat. Rev. Genet . 3 (6): 415–28 . doi :10.1038/nrg816. PMID 12042769. S2CID 2122000.
- ^ Costello JF, Frühwald MC, Smiraglia DJ и др. (февраль 2000 г.). «Аберрантное метилирование CpG-островков имеет неслучайные и специфичные для типа опухоли паттерны». Nat. Genet . 24 (2): 132– 8. doi :10.1038/72785. PMID 10655057. S2CID 6273676.
- ^ Zardo G, Tiirikainen MI, Hong C и др. (ноябрь 2002 г.). «Интегрированные геномные и эпигеномные анализы выявляют инактивацию биаллельных генов в опухолях». Nat. Genet . 32 (3): 453– 8. doi :10.1038/ng1007. PMID 12355068. S2CID 23645606.
- ^ Yu L, Liu C, Vandeusen J, et al. (март 2005 г.). «Глобальная оценка метилирования промотора в мышиной модели рака идентифицирует ID4 как предполагаемый ген-супрессор опухолей при лейкемии человека». Nat. Genet . 37 (3): 265–74 . doi :10.1038/ng1521. PMID 15723065. S2CID 515916.
- ^ Хатада И, Хаяшизаки И, Хироцунэ С, Комацубара Х, Мукаи Т (ноябрь 1991 г.). «Метод геномного сканирования высших организмов с использованием сайтов рестрикции в качестве ориентиров». Proc. Natl. Acad. Sci. USA . 88 (21): 9523– 7. Bibcode :1991PNAS...88.9523H. doi : 10.1073/pnas.88.21.9523 . PMC 52750 . PMID 1946366.
- ^ Ракян В.К., Хильдманн Т., Новик КЛ. и др. (декабрь 2004 г.). «Профилирование метилирования ДНК главного комплекса гистосовместимости человека: пилотное исследование для проекта человеческого эпигенома». PLOS Biol . 2 (12): e405. doi : 10.1371/journal.pbio.0020405 . PMC 529316. PMID 15550986 .

- ^ Gitan RS, Shi H, Chen CM, Yan PS, Huang TH (январь 2002 г.). «Methylation-Specific Oligonucleotide Microarray: A New Potential for High-Throughput Methylation Analysis». Genome Res . 12 (1): 158– 64. doi :10.1101/gr.202801. PMC 155260. PMID 11779841 .
- ^ Meissner A, Gnirke A, Bell GW, Ramsahoye B, Lander ES, Jaenisch R (2005). "Секвенирование бисульфита с уменьшенным представлением для сравнительного анализа метилирования ДНК с высоким разрешением". Nucleic Acids Res . 33 (18): 5868– 77. doi :10.1093/nar/gki901. PMC 1258174. PMID 16224102 .
- ^ Adorján P, Distler J, Lipscher E и др. (март 2002 г.). «Прогнозирование и обнаружение класса опухолей с помощью анализа метилирования ДНК на основе микрочипов». Nucleic Acids Res . 30 (5): 21e–21. doi :10.1093/nar/30.5.e21. PMC 101257. PMID 11861926 .
- ^ Dai Z, Weichenhan D, Wu YZ и др. (октябрь 2002 г.). «Библиотека границ AscI для исследований генетических и эпигенетических изменений в CpG-островках». Genome Res . 12 (10): 1591– 8. doi :10.1101/gr.197402. PMC 187524. PMID 12368252 .
- ^ abcd Pomraning KR, Smith KM, Freitag M (март 2009). "Высокопроизводительный анализ метилирования ДНК в эукариотах по всему геному". Методы . 47 (3): 142– 50. doi :10.1016/j.ymeth.2008.09.022. PMID 18950712.
- ^ abcd Down TA, Rakyan VK, Turner DJ и др. (июль 2008 г.). «Стратегия байесовской деконволюции для анализа метилома ДНК на основе иммунопреципитации». Nat. Biotechnol . 26 (7): 779– 85. doi :10.1038/nbt1414. PMC 2644410. PMID 18612301 .
- ^ ab Jacinto FV, Ballestar E, Esteller M (январь 2008 г.). «Иммунопреципитация метил-ДНК (MeDIP): поиск метилома ДНК». BioTechniques . 44 (1): 35– 43. doi : 10.2144/000112708 . PMID 18254377.
- ^ Meehan RR, Lewis JD, Bird AP (октябрь 1992 г.). «Характеристика MeCP2, белка связывания ДНК позвоночных с аффинностью к метилированной ДНК». Nucleic Acids Res . 20 (19): 5085–92 . doi :10.1093/nar/20.19.5085. PMC 334288. PMID 1408825 .
- ^ ab Wilson IM, et al. (2005). "Эпигеномика: картирование метилома". Cell Cycle . 5 (2): 155– 8. doi : 10.4161/cc.5.2.2367 . PMID 16397413.
- ^ abc Zhang X, Yazaki J, Sundaresan A, et al. (сентябрь 2006 г.). «Полногеномное картирование высокого разрешения и функциональный анализ метилирования ДНК у арабидопсиса». Cell . 126 (6): 1189– 201. doi : 10.1016/j.cell.2006.08.003 . PMID 16949657.
- ^ Микрочип метилирования ДНК
- ^ «Illumina | Решения на основе секвенирования и матриц для генетических исследований».
- ^ "Секвенирование ДНК, скрининг и анализ: приборы и услуги - 454 Life Sciences™". Архивировано из оригинала 2008-03-18 . Получено 2008-03-18 .
- ^ Li H, Ruan J, Durbin R (ноябрь 2008 г.). «Картирование коротких прочтений секвенирования ДНК и вызов вариантов с использованием оценок качества картирования». Genome Res . 18 (11): 1851– 8. doi :10.1101/gr.078212.108. PMC 2577856. PMID 18714091 .
- ^ Esteller M (апрель 2007 г.). «Эпигенетическое подавление генов при раке: гиперметилом ДНК». Hum. Mol. Genet . 16 (Spec No 1): R50–9. doi : 10.1093/hmg/ddm018 . PMID 17613547.
- ^ Geeleher P, Hartnett L, Egan LJ, Golden A, Raja Ali RA, Seoighe C (июнь 2013 г.). «Анализ набора генов сильно смещен при применении к данным метилирования по всему геному». Bioinformatics . 29 (15): 1851– 7. doi : 10.1093/bioinformatics/btt311 . PMID 23732277.
- ^ Кешет И, Шлезингер И, Фаркаш С и др. (февраль 2006 г.). «Доказательства поучительного механизма метилирования de novo в раковых клетках». Nat. Genet . 38 (2): 149–53 . doi :10.1038/ng1719. PMID 16444255. S2CID 14405389.
- ^ Novak P, Jensen T, Oshiro MM и др. (ноябрь 2006 г.). «Эпигенетическая инактивация кластера генов HOXA при раке груди». Cancer Res . 66 (22): 10664– 70. doi : 10.1158/0008-5472.CAN-06-2761 . PMID 17090521.
