РНК-носитель-трансфер
| РНК-носитель-трансфер | |
|---|---|
 РНК-носитель ( PDB : 3IYR ) | |
| Идентификаторы | |
| Символ | тмРНК |
| Рфам | РФ00023 |
| Другие данные | |
| Тип РНК | ген |
| Структуры PDB | ПДБе |
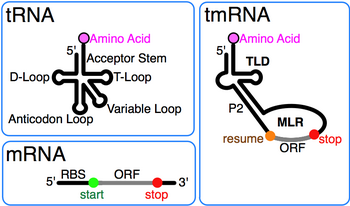
Трансферная мРНК (сокращенно тмРНК , также известная как 10Sa РНК и по своему генетическому названию SsrA ) представляет собой бактериальную молекулу РНК с двойными свойствами тРНК -подобными и матричной РНК- подобными. ТмРНК образует рибонуклеопротеиновый комплекс ( тмРНП ) вместе с малым белком B (SmpB), фактором элонгации Tu ( EF-Tu ) и рибосомальным белком S1. При транс -трансляции тмРНК и связанные с ней белки связываются с бактериальными рибосомами , которые остановились в середине биосинтеза белка , например, при достижении конца матричной РНК, которая потеряла свой стоп-кодон. ТмРНК удивительно универсальна: она перерабатывает остановившуюся рибосому, добавляет метку, индуцирующую протеолиз, к незавершенному полипептиду и облегчает деградацию аберрантной матричной РНК . [1] У большинства бактерий эти функции выполняются стандартными однокомпонентными тмРНК. У других видов бактерий пермутированный ген ssrA производит двухкомпонентную тмРНК, в которой две отдельные цепи РНК соединены спариванием оснований.
Открытие и ранние работы
tmRNA была впервые обозначена как 10Sa РНК в 1979 году после того, как смешанная электрофоретическая фракция "10S" РНК Escherichia coli была далее разделена на tmRNA и РНК РНКазы P аналогичного размера (10Sb). [2] Присутствие псевдоуридина в смешанной 10S РНК намекало на то, что tmRNA имеет модифицированные основания, обнаруженные также в tRNA . Сходство 3'-конца tmRNA с петлей T tRNA было впервые обнаружено при секвенировании ssrA из Mycobacterium tuberculosis . [3] Последующее сравнение последовательностей выявило полный tRNA-подобный домен (TLD), образованный 5'- и 3'- концами tmRNA, включая акцепторный стебель с элементами, подобными тем, что находятся в аланиновой tRNA, которые способствуют ее аминоацилированию аланин-tRNA-лигазой . [4] Также были выявлены отличия от тРНК : в тмРНК отсутствует антикодоновая часть, а область D-части представляет собой петлю без пар оснований.
Структура
Вторичная структура стандартных однокомпонентных тмРНК

Полная вторичная структура тмРНК E. coli была выяснена с помощью сравнительного анализа последовательностей и структурного зондирования . [5] [6] Пары оснований Уотсона-Крика и GU были идентифицированы путем сравнения последовательностей бактериальной тмРНК с использованием автоматизированных вычислительных методов в сочетании с ручными процедурами выравнивания . [7] [8] На прилагаемом рисунке показана схема спаривания оснований этой прототипической тмРНК, которая организована в 12 филогенетически поддерживаемых спиралей (также называемых парами P1–P12), некоторые из которых разделены на спиральные сегменты.
Характерной особенностью каждой тмРНК является консервативный тРНК-подобный домен (TLD), состоящий из спиралей 1, 12 и 2a (аналоги акцепторного стебля тРНК, Т-стебля и вариабельного стебля соответственно) и содержащий 5' монофосфат и аланилируемый 3' CCA-концы. МРНК-подобный регион (MLR) в стандартной тмРНК представляет собой большую петлю, содержащую псевдоузлы и кодирующую последовательность (CDS) для тегового пептида , отмеченную кодоном резюме и стоп-кодоном . Кодируемый теговый пептид (ANDENYALAA в E. coli ) различается среди бактерий, возможно, в зависимости от набора доступных протеаз и адаптеров. [9]
tmRNAs обычно содержат четыре псевдоузла , один (pk1) выше по течению от пептида-тега CDS, и другие три псевдоузла (pk2 - pk4) ниже по течению от CDS. Области псевдоузлов, хотя в целом консервативны, являются эволюционно пластичными. Например, в (цельных) tmRNAs цианобактерий pk4 заменен двумя тандемно расположенными меньшими псевдоузлами. Это говорит о том, что сворачивание tmRNA вне TLD может быть важным, однако в области псевдоузла отсутствуют консервативные остатки, и псевдоузлы являются одними из первых структур, которые теряются, когда последовательности ssrA расходятся в пластидных и эндосимбионтных линиях. Спаривание оснований в области из трех псевдоузлов tmRNA E. coli нарушается во время транс-трансляции. [7] [10]
Двухкомпонентные тмРНК
Циркулярно переставленный ssrA был зарегистрирован в трех основных линиях: i) все альфапротеобактерии и примитивные митохондрии якобидных протистов, ii) две разрозненные группы цианобактерий ( Gloeobacter и клада, содержащая Prochlorococcus и многие Synechococcus ), и iii) некоторые члены бетапротеобактерий ( Cupriavidus и некоторые Rhodocyclales). [11] [12] Все они производят одну и ту же общую двухкомпонентную (акцепторную и кодирующую части) форму, эквивалентную стандартной форме, надрезанной ниже рамки считывания. Ни одна из них не сохраняет более двух псевдоузлов по сравнению с четырьмя (или более) стандартной тмРНК.
У альфапротеобактерий есть две характерные последовательности: замена типичной последовательности T-петли TΨCRANY на GGCRGUA и последовательность AACAGAA в большой петле 3´-концевого псевдоузла. В митохондриях MLR утрачен, а примечательная перестановка митохондриального ssrA приводит к небольшому цельному продукту в Jakoba libera . [13]
Цианобактерии представляют собой наиболее правдоподобный случай эволюции пермутированного гена из стандартного гена из-за значительного сходства последовательностей между двумя типами генов, которые встречаются в различных штаммах Synechococcus .
процессинг тмРНК
Большинство тмРНК транскрибируются как более крупные предшественники, которые обрабатываются во многом подобно тРНК . Расщепление на 5´ конце осуществляется рибонуклеазой P. [ 4] В процессинге 3´ конца тмРНК могут участвовать несколько экзонуклеаз, хотя наиболее эффективны РНКаза T и РНКаза PH . [14] [15] В зависимости от вида бактерий 3'-CCA либо кодируется, либо добавляется нуклеотидилтрансферазой тРНК .
Подобная обработка на внутренних участках пермутированной предшественника tmRNA объясняет ее физическое разделение на две части. Двухкомпонентные tmRNA имеют два дополнительных конца, обработку которых необходимо учитывать. Для альфапротеобактерий один 5´ конец является необработанным стартовым сайтом транскрипции. [16] Дальний 3´ конец может в некоторых случаях быть результатом rho-независимого завершения.
Трехмерные структуры


Структуры с высоким разрешением полных молекул tmRNA в настоящее время недоступны и могут быть труднодоступны из-за присущей MLR гибкости. В 2007 году кристаллическая структура TLD Thermus thermophilus, связанного с белком SmpB, была получена с разрешением 3 Å. Эта структура показывает, что SmpB имитирует стебель D и антикодон канонической tRNA, тогда как спиральный участок 2a tmRNA соответствует вариабельному плечу tRNA. [18] Исследование tmRNA с помощью криоэлектронной микроскопии на ранней стадии транс-трансляции показывает пространственную связь между рибосомой и tmRNP (tmRNA, связанная с белком EF-Tu ). TLD расположен вблизи центра, связанного с ГТФазой, в 50S рибосомальной субъединице; Спираль 5 и псевдоузлы pk2-pk4 образуют дугу вокруг клюва рибосомальной субъединицы 30S. [19]
Транс-перевод

Кодирование tmRNA было обнаружено в 1995 году [20] , когда Симпсон и его коллеги сверхэкспрессировали цитокин мыши IL-6 в E. coli и обнаружили несколько укороченных пептидов , полученных из цитокина , каждый из которых был помечен на карбоксильных концах тем же самым 11-аминокислотным остатком расширения (A)ANDENYALAA. За исключением N-концевого аланина , который исходит из 3'-конца самой tmRNA, эта последовательность тега была прослежена до короткой открытой рамки считывания в tmRNA E. coli . Кейлер и др. признали, что пептид тега обеспечивает протеолиз , и предложили модель транс -трансляции для действия tmRNA. [21]
В то время как детали механизма транс -трансляции находятся в стадии изучения, в целом принято считать, что tmRNA сначала занимает пустой сайт A остановившейся рибосомы . Затем рибосома перемещается с 3'-конца укороченной информационной РНК на кодон возобновления MLR, за которым следует стадия, склонная к проскальзыванию, с которой трансляция продолжается нормально до тех пор, пока не встретится стоп-кодон tmRNA в рамке считывания. Транс-трансляция необходима для некоторых видов бактерий, тогда как другим бактериям tmRNA требуется для выживания при воздействии стрессовых условий роста. [22] Считается, что tmRNA может помочь клетке с устойчивостью к антибиотикам, спасая рибосомы, остановившиеся из-за антибиотиков. [23] В зависимости от организма, пептид-метка может распознаваться различными протеазами или адаптерами протеаз. [9]
Мобильные генетические элементы и ген тмРНК

ssrA является как целью для некоторых мобильных ДНК, так и пассажиром на других. Было обнаружено, что он прерывается тремя типами мобильных элементов. При помощи различных стратегий ни один из них не нарушает функцию гена: интроны группы I удаляют себя путем самосплайсинга, риккетсиальные палиндромные элементы (RPE) вставляются в безвредные сайты, а геномные острова, кодирующие интегразу, разделяют свой целевой ssrA , но восстанавливают отщепленную часть. [24] [25] [26] [27]
Нехромосомный ssrA был впервые обнаружен в геномном исследовании микобактериофагов (у 10% фагов). [28] Другие мобильные элементы , включая плазмиды и геномные острова, были обнаружены с ssrA . Одним из интересных случаев является Rhodobacter sphaeroides ATCC 17025, чей нативный ген tmRNA нарушен геномным островом; в отличие от всех других геномных островов в генах tmRNA (или tRNA), этот остров инактивировал нативный целевой ген без восстановления, но компенсирует это, неся свой собственный ген tmRNA. Очень необычный родственник ssrA обнаружен в литическом микобактериофаге DS6A, который кодирует немного больше, чем TLD.
Митохондриальные тмРНК (ссрАген)
Митохондриально-кодируемая, структурно редуцированная форма тмРНК (мт-тмРНК) была впервые постулирована для жгутиконосца якобид Reclinomonas americana . [11] Впоследствии наличие митохондриального гена ( ssrA ), кодирующего тмРНК, а также сайтов транскрипции и процессинга РНК были подтверждены для всех, кроме одного представителя якобид . [29] [13] Функциональные доказательства, то есть аминоацилирование мт- тмРНК аланином , доступны для Jakoba libera . [13] Совсем недавно ssrA был также идентифицирован в митохондриальных геномах оомицетов . [30] Как и в α-Proteobacteria (предках митохондрий ), мт-тмРНК представляют собой циклически переставленные двухкомпонентные молекулы РНК, за исключением Jakoba libera , где ген вернулся к кодированию однокомпонентной конформации тмРНК. [13]
ИдентификацияссрАв митохондриальных геномах

Гены митохондриальной tmRNA изначально были распознаны как короткие последовательности, которые сохраняются среди якобид и которые имеют потенциал для складывания в отчетливую вторичную структуру, подобную тРНК. С доступностью девяти полных последовательностей мтДНК якобид [29] и значительно улучшенным инструментом поиска ковариации (Infernal; [31] [32] [33] ), была разработана модель ковариации на основе митохондриальных tmRNA якобид , которая идентифицировала митохондриальные гены ssrA также у оомицетов . В настоящее время обнаружено в общей сложности 34 мт-tmRNA оомицетов в шести родах: Albugo , Bremia , Phytophthora , Pseudoperonospora , Pythium и Saprolegnia . Ковариационная модель, созданная с использованием последовательностей как якобид , так и оомицетов, теперь доступна на сайте Rfam под названием «mt-tmRNA». [30]
Структура мт-тРНК
Стандартная бактериальная тмРНК состоит из тРНК(Ала)-подобного домена (позволяющего добавлять некодируемый аланин к мРНК, у которых отсутствует стоп-кодирование), и мРНК-подобного домена, кодирующего белковую метку, которая направляет полипептид на протеолиз. МРНК-подобный домен был утрачен в мт-тмРНК. Сравнительный анализ последовательностей указывает на особенности, типичные для мт-тмРНК. [30] Наиболее консервативной является первичная последовательность аминоацильного акцепторного стебля. Эта часть молекулы имеет неизменный остаток А в позиции дискриминатора и пару GU в позиции 3 (за исключением S eculamonas ecuadoriensis , у которой есть пара GC); эта позиция является сайтом распознавания для аланил-тРНК-синтазы. P2 представляет собой спираль переменной длины (от 3 до 10 пар оснований) и соответствует антикодоновому стеблю тРНК, но без антикодоновой петли (так как она не требуется для функции тмРНК). P2 стабилизирует тРНК-подобную структуру, но четыре нуклеотида, инвариантные у оомицетов и якобид, предполагают дополнительную, в настоящее время не идентифицированную функцию. P3 имеет пять пар оснований и соответствует Т-плечу тРНК, но с различными консенсусными нуклеотидами как в парной области, так и в петле. Последовательность Т-петли сохраняется у оомицетов и якобид , с небольшими отклонениями (например, Saprolegnia ferax ). Наконец, вместо тРНК-подобного D-стебля с укороченной трехнуклеотидной D-петлей, характерной для бактериальных тмРНК, митохондриальные аналоги имеют высоковариабельную петлю длиной от 5 до 14 нуклеотидов. Промежуточная последовательность (Int.) двухкомпонентных мт-тРНК богата A+U и имеет нерегулярную длину (4-34 нт). Модели вторичной структуры одно- и двухкомпонентных мт-тРНК см. на рисунке 1.
Обработка и экспрессия мт-тРНК

Данные РНК-секвенирования Phytophthora sojae показывают уровень экспрессии, аналогичный уровню соседних митохондриальных тРНК , а четыре основных участка процессинга подтверждают предсказанные концы зрелой мт-тРНК. [30] Молекула-предшественник тмРНК, вероятно, процессируется РНКазой P и эндонуклеазой процессинга тРНК 3' (см. Рисунок 2); предполагается, что последняя активность приводит к удалению промежуточной последовательности. После добавления CCA к нуклеотиду-дискриминатору 3' тмРНК может быть заряжена аланил-тРНК-синтетазой с аланином.
Смотрите также
Ссылки
- ^ Keiler KC (2008). «Биология транс-трансляции». Annual Review of Microbiology . 62 : 133–51 . doi :10.1146/annurev.micro.62.081307.162948. PMID 18557701.
- ^ Ray BK, Apirion D (июль 1979). «Характеристика 10S РНК: новая стабильная молекула РНК из Escherichia coli». Molecular & General Genetics . 174 (1): 25– 32. doi :10.1007/BF00433301. PMID 384159. S2CID 22699560.
- ^ Tyagi JS, Kinger AK (январь 1992). "Идентификация структурного гена 10Sa РНК Mycobacterium tuberculosis". Nucleic Acids Research . 20 (1): 138. doi : 10.1093 /nar/20.1.138. PMC 310338. PMID 1371186.
- ^ ab Komine Y, Kitabatake M, Yokogawa T, Nishikawa K, Inokuchi H (сентябрь 1994 г.). «Структура, подобная тРНК, присутствует в 10Sa РНК, небольшой стабильной РНК из Escherichia coli». Труды Национальной академии наук Соединенных Штатов Америки . 91 (20): 9223– 7. Bibcode : 1994PNAS...91.9223K. doi : 10.1073/pnas.91.20.9223 . PMC 44784. PMID 7524073 .
- ^ Уильямс КП, Бартель ДП (декабрь 1996 г.). «Филогенетический анализ вторичной структуры тРНК». РНК . 2 (12): 1306–10 . PMC 1369456. PMID 8972778 .
- ^ Фельден Б., Химено Х., Муто А., Маккатчеон Дж. П., Аткинс Дж. Ф., Гестеланд РФ (январь 1997 г.). «Исследование структуры 10Sa РНК (тмРНК) Escherichia coli». РНК . 3 (1): 89–103 . ПМЦ 1369465 . ПМИД 8990402.
- ^ ab Zwieb C, Wower I, Wower J (май 1999). "Сравнительный анализ последовательностей тРНК". Nucleic Acids Research . 27 (10): 2063– 71. doi :10.1093/nar/27.10.2063. PMC 148424. PMID 10219077 .
- ^ Андерсен Э.С., Линд-Томсен А., Кнудсен Б., Кристенсен С.Е., Хавгаард Дж.Х., Тораринссон Э., Ларсен Н., Цвиб С., Сестофт П., Кьемс Дж., Городкин Дж. (ноябрь 2007 г.). «Полуавтоматическое улучшение выравнивания РНК». РНК . 13 (11): 1850–9 . doi :10.1261/rna.215407. ПМК 2040093 . ПМИД 17804647.
- ^ ab Gur E, Sauer RT (октябрь 2008 г.). «Эволюция тега деградации ssrA в микоплазме: переключение специфичности на другую протеазу». Труды Национальной академии наук Соединенных Штатов Америки . 105 (42): 16113– 8. Bibcode : 2008PNAS..10516113G . doi : 10.1073/pnas.0808802105 . PMC 2570983. PMID 18852454.
- ^ Wower IK, Zwieb C, Wower J (май 2005 г.). «Трансферная РНК разворачивается при прохождении рибосомы». РНК . 11 (5): 668– 73. doi :10.1261/rna.7269305. PMC 1370753 . PMID 15811920.
- ^ ab Keiler KC, Shapiro L, Williams KP (июль 2000 г.). «tmRNAs, которые кодируют теги, индуцирующие протеолиз, обнаружены во всех известных бактериальных геномах: двухкомпонентная tmRNA функционирует у Caulobacter». Труды Национальной академии наук Соединенных Штатов Америки . 97 (14): 7778– 83. Bibcode :2000PNAS...97.7778K. doi : 10.1073/pnas.97.14.7778 . PMC 16621 . PMID 10884408.
- ^ Sharkady SM, Williams KP (2004). «Третья линия с двухкомпонентной тмРНК». Nucleic Acids Research . 32 (15): 4531– 8. doi : 10.1093/nar/gkh795. PMC 516066. PMID 15326226.
- ^ abcd Jacob Y, Seif E, Paquet PO, Lang BF (апрель 2004 г.). «Утрата мРНК-подобной области в митохондриальных тмРНК якобидов». РНК . 10 (4): 605– 14. doi :10.1261 / rna.5227904. PMC 1370551. PMID 15037770.
- ^ Шривастава РА, Шривастава Н, Апирион Д (май 1992). «Характеристика фермента РНК-процессинга РНКазы III из дикого типа и сверхэкспрессирующих клеток Escherichia coli при обработке природных субстратов РНК». Международный журнал биохимии . 24 (5): 737– 49. doi :10.1016/0020-711X(92)90007-N. PMID 1375563.
- ^ Li Z, Pandit S, Deutscher MP (март 1998 г.). «3'-экзорибонуклеолитическая обрезка — общая черта созревания малых стабильных РНК в Escherichia coli». Труды Национальной академии наук Соединенных Штатов Америки . 95 (6): 2856– 61. Bibcode : 1998PNAS...95.2856L. doi : 10.1073 /pnas.95.6.2856 . PMC 19659. PMID 9501180.
- ^ Mao C, Bhardwaj K, Sharkady SM, Fish RI, Driscoll T, Wower J, Zwieb C, Sobral BW, Williams KP (2009). «Вариации гена tmRNA». RNA Biology . 6 (4): 355–61 . doi : 10.4161/rna.6.4.9172 . PMID 19617710.
- ^ Someya T, Nameki N, Hosoi H, Suzuki S, Hatanaka H, Fujii M, Terada T, Shirouzu M, Inoue Y, Shibata T, Kuramitsu S, Yokoyama S, Kawai G (январь 2003 г.). "Структура раствора белка, связывающего tmRNA, SmpB, из Thermus thermophilus". FEBS Letters . 535 ( 1– 3): 94– 100. Bibcode : 2003FEBSL.535...94S. doi : 10.1016/S0014-5793(02)03880-2 . PMID 12560085.
- ^ ab Bessho Y, Shibata R, Sekine S, Murayama K, Higashijima K, Hori-Takemoto C, Shirouzu M, Kuramitsu S, Yokoyama S (май 2007 г.). "Структурная основа функциональной мимикрии длинной вариабельной тРНК с помощью РНК-переносчика". Труды Национальной академии наук Соединенных Штатов Америки . 104 (20): 8293– 8. Bibcode : 2007PNAS..104.8293B . doi : 10.1073/pnas.0700402104 . PMC 1895943. PMID 17488812.
- ^ Valle M, Gillet R, Kaur S, Henne A, Ramakrishnan V, Frank J (апрель 2003 г.). «Визуализация входа тмРНК в остановившуюся рибосому». Science . 300 (5616): 127– 30. Bibcode :2003Sci...300..127V. doi :10.1126/science.1081798. PMID 12677067. S2CID 28845151.
- ^ Tu GF, Reid GE, Zhang JG, Moritz RL, Simpson RJ (апрель 1995 г.). «C-концевое расширение укороченных рекомбинантных белков в Escherichia coli с 10Sa РНК-декапептидом». Журнал биологической химии . 270 (16): 9322– 6. doi : 10.1074/jbc.270.16.9322 . PMID 7536743.
- ^ Keiler KC, Waller PR, Sauer RT (февраль 1996 г.). «Роль системы пептидной маркировки в деградации белков, синтезированных из поврежденной информационной РНК». Science . 271 (5251): 990– 3. Bibcode :1996Sci...271..990K. doi :10.1126/science.271.5251.990. PMID 8584937. S2CID 29254050.
- ^ Thibonnier M, Thiberge JM, De Reuse H (2008). Ahmed N (ред.). "Транс-трансляция в Helicobacter pylori: важность спасения рибосомы и необходимость маркировки белка для устойчивости к стрессу и компетентности". PLOS ONE . 3 (11): e3810. Bibcode :2008PLoSO...3.3810T. doi : 10.1371/journal.pone.0003810 . PMC 2584231 . PMID 19043582.
- ^ Мюллер, Клаудия; Кроу-Маколиф, Кайлан; Уилсон, Дэниел Н. (18.03.2021). «Пути спасения рибосом у бактерий». Frontiers in Microbiology . 12 : 652980. doi : 10.3389/fmicb.2021.652980 . ISSN 1664-302X. PMC 8012679. PMID 33815344 .
- ^ Kirby JE, Trempy JE, Gottesman S (апрель 1994 г.). «Иссечение криптического профага, похожего на P4, приводит к экспрессии протеазы Alp в Escherichia coli». Journal of Bacteriology . 176 (7): 2068– 81. doi :10.1128/jb.176.7.2068-2081.1994. PMC 205313 . PMID 7511583.
- ^ Williams KP (январь 2002 г.). «Сайт tmRNA: вторжение интрона». Nucleic Acids Research . 30 (1): 179– 82. doi :10.1093/nar/30.1.179. PMC 99078. PMID 11752287 .
- ^ Дуайер DS (январь 2001). «Эгоистичная ДНК и происхождение генов». Science . 291 (5502): 252– 3. doi :10.1126/science.291.5502.252. PMID 11253208. S2CID 5369275.
- ^ Williams KP (февраль 2003 г.). «Трафик в гене tmRNA». Журнал бактериологии . 185 (3): 1059–70 . doi :10.1128/JB.185.3.1059-1070.2003. PMC 142792. PMID 12533482 .
- ^ Hatfull GF, Pedulla ML, Jacobs-Sera D, Cichon PM, Foley A, Ford ME, Gonda RM, Houtz JM, Hryckowian AJ, Kelchner VA, Namburi S, Pajcini KV, Popovich MG, Schleicher DT, Simanek BZ, Smith AL, Zdanowicz GM, Kumar V, Peebles CL, Jacobs WR, Lawrence JG, Hendrix RW (июнь 2006 г.). «Изучение метапротеома микобактериофага: геномика фага как образовательная платформа». PLOS Genetics . 2 (6): e92. doi : 10.1371/journal.pgen.0020092 . PMC 1475703. PMID 16789831 .
- ^ ab Burger G, Gray MW, Forget L, Lang BF (2013). «Поразительно бактериоподобные и богатые генами митохондриальные геномы среди простейших якобид». Genome Biology and Evolution . 5 (2): 418–38 . doi :10.1093/gbe/evt008. PMC 3590771. PMID 23335123 .
- ^ abcd Hafez M, Burger G, Steinberg SV, Lang BF (июль 2013 г.). «Вторая эукариотическая группа с кодируемой митохондриями тмРНК: идентификация in silico и экспериментальное подтверждение». RNA Biology . 10 (7): 1117– 24. doi :10.4161/rna.25376. PMC 3849159 . PMID 23823571. Архивировано из оригинала 21.02.2014 . Получено 13.02.2014 .
- ^ Эдди, С. "Адский веб-сайт" . Получено 14 августа 2016 г.
- ^ Эдди SR, Дурбин Р. (июнь 1994 г.). «Анализ последовательности РНК с использованием ковариационных моделей». Nucleic Acids Research . 22 (11): 2079– 88. doi :10.1093/nar/22.11.2079. PMC 308124. PMID 8029015 .
- ^ Nawrocki EP, Kolbe DL, Eddy SR (май 2009). «Infernal 1.0: вывод выравниваний РНК». Биоинформатика . 25 (10): 1335– 7. doi :10.1093/bioinformatics/btp157. PMC 2732312. PMID 19307242 .
Дальнейшее чтение
Внешние ссылки
- tmRDB: База данных последовательностей tmRNA
- Сайт тмРНК
- Запись Rfam для tmRNA [ постоянная мертвая ссылка ]