DEPDC5
| DEPDC5 | |||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Идентификаторы | |||||||||||||||||||||||||||||||||||||||||||||||||||
| Псевдонимы | DEPDC5 , DEP.5, FFEVF, домен DEP, содержащий 5, FFEVF1, домен DEP, содержащий 5, субъединица подкомплекса GATOR1 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| Внешние идентификаторы | ОМИМ : 614191; МГИ : 2141101; гомологен : 34718; GeneCards : DEPDC5; OMA :DEPDC5 — ортологи | ||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Викиданные | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
DEPDC5 (или домен DEP, содержащий 5 ) — это человеческий белок с плохо изученной функцией, но в нескольких исследованиях его связывали с раком . [5] [6] Он кодируется геном с таким же названием, расположенным на хромосоме 22 .
Функция
Функция DEPDC5 пока неизвестна, но предполагается, что она участвует во внутриклеточной передаче сигнала на основе гомологии между доменами DEP DEPDC5 и Dishevelled-1 ( DVL1 ). [7]
Мутации в этом гене связаны со случаями фокальной эпилепсии (doi:10.1038/ng.2601).
Ген
У Homo sapiens ген DEPDC5 локализован на длинном плече хромосомы 22, 22q12.2-q12.3, между генами PRRL14 и YWHAH . Клиническая значимость этого гена включает интронный SNP (rs1012068), который был связан с 2-кратным увеличением риска гепатоцеллюлярной карциномы . [5]

Структура
Домены

ДЕП
Домен DEP получил свое название от белков Dishevelled , Egl-10 и Pleckstrin , каждый из которых содержит вариант этого домена. [8] Он охватывает 82 остатка и составляет 343 аминокислоты от C-конца . SWISS-MODEL предсказывает два бета-слоя и три альфа-спирали, содержащиеся в домене. [9]
Хотя его точная функция неизвестна, домен DEPDC5 DEP имеет наибольшее структурное сходство с доменом DEP DVL1 при выполнении CBLAST в NCBI . [10] Выравнивание оценивается как Evalue 1.00e-08 и указывает на 30% идентичности между доменами DEP двух белков. В DVL1 домен DEP участвует в локализации белка на плазматической мембране как часть сигнального пути Wnt . [11]
ДУФ 3608
Домен DUF 3608 находится в 99 аминокислотах от N-конца и сам охватывает 280 аминокислот. PELE предсказывает по крайней мере один бета-лист и две альфа-спирали в пределах этого домена. [12] Он также содержит 26 высококонсервативных остатков и несколько посттрансляционных модификаций. Оба случая рассматриваются далее в этой статье.
Доказательства функции DUF 3608 были обнаружены в дрожжевом гомологе Iml1p. Считается, что DUF 3608 Imlp1 помогает связываться с двумя белками-партнерами, Npr2 и Npr3. Вместе эти три белка образуют комплекс Iml1-Npr2-Npr3 и участвуют в регуляции аутофагии «неазотного голодания» . Исследователи, которые это обнаружили, предлагают переименовать DUF 3608 в RANS (Required for Autophagy induced under Non-nitrogen Starvation conditions). [13]
Вторичная структура
На основе единогласного консенсуса инструмента прогнозирования вторичной структуры PELE, DEPDC5 содержит по крайней мере десять альфа-спиралей и девять бета-слоев. Расположение этих вторичных структур показано на изображении ниже: красные выделения — это альфа-спирали, а синие выделения — это бета-слои.

Гомология
Ортологи
Грибы являются наиболее отдаленно родственными организмами, содержащими белок, ортологичный человеческому DEPDC5, включая Saccharomyces cerevisiae и Albugo laibachii . У грибов название белка Iml1p или вакуолярный мембранно-ассоциированный белок Iml1. Отклонения в названии у других организмов включают CG12090 ( дрозофила ) и AGAP007010 ( комар ). [7] Консервация высока между людьми и другими видами позвоночных , варьируясь от 74% идентичности у цихлид до 99% идентичности у шимпанзе . [14]
В следующей таблице представлен анализ 20 белков, ортологичных человеческому DEPDC5.
| Разновидность | Общее название | Регистрационный номер NCBI | Имя NCBI | Длина | Идентификация последовательности | Сходство последовательностей | Годы с момента расхождения с человеком (млн. лет назад) [15] |
|---|---|---|---|---|---|---|---|
| Пан троглодиты | Шимпанзе | XP_003317262 | DEPDC5 | 1572 г. до н.э. | 99% | 99% | 6.4 |
| Nomascus leucogenys | Гиббон | XP_003258163 | DEPDC5 | 1602 г. до н.э. | 99% | 99% | 20.4 |
| Mus musculus | Мышь | NP_001164038 | DEPDC5 | 1591 г. до н.э. | 94% | 96% | 92.4 |
| Бос Таурус | Корова | XP_002694678 | DEPDC5 | 1593 г. до н.э. | 94% | 96% | 94.4 |
| Sorex araneus | Землеройка | ACE77702 | DEPDC5 | 1570 г. до н.э. | 94% | 96% | 94.4 |
| Монодельфис домашний | Опоссум | XP_001378772 | DEPDC5 | 1522 г. до н.э. | 89% | 93% | 163,9 |
| Галлус галлус | Курица | XP_415249 | DEPDC5 | 1592 г. до н.э. | 88% | 93% | 301.7 |
| Meleagris gallopavo | Турция | XP_003211073 | DEPDC5 | 1592 г. до н.э. | 88% | 93% | 301.7 |
| Taeniopygia guttata | Зебровая амадина | XP_002199825 | DEPDC5 | 1572 г. до н.э. | 87% | 92% | 301.7 |
| Xenopus tropicalis | Лягушка | XP_002931964 | DEPDC5-подобный | 1574 г. до н.э. | 79% | 86% | 371.2 |
| Данио рерио | Рыба-зебра | XP_691450 | DEPDC5-подобный | 1590 г. до н.э. | 75% | 84% | 400.1 |
| Ореохромис нилотический | Цихлида | XP_003459226 | DEPDC5 | 1577 г. до н.э. | 74% | 82% | 400.1 |
| Strongylocentrotus purpuratus | Морской ёж | XP_794020 | похоже на DEPDC5 | 1608 г. до н.э. | 43% | 57% | 742.9 |
| Дрозофила меланогастер | Дрозофила | NP_647618 | GC12090 | 1471 год до нашей эры | 41% | 57% | 782.7 |
| Педикулюс человеческий корпорис | Вошь | XP_002429401 | DEPDC, предполагаемый | 1538 г. до н.э. | 38% | 53% | 782.7 |
| Anopheles gambiae | Комар | XP_308760 | AGAP007010-PA | 1640 г. до н.э. | 36% | 51% | 782.7 |
| Ascaris suum | аскарида | ADY40551 | DEPDCp5 | 1359 аа | 31% | 51% | 937,5 |
| Ustilago maydis | Головня кукурузы | XP_757759 | вакуолярно-ассоциированный белок Iml1 | 1867 г. до н.э. | 23% | 52% | 1215.8 |
| Сахаромицеты cerevisiae | Дрожжи | NP_012672 | Iml1p | 1584 г. до н.э. | 20% | 50% | 1215.8 |
| Альбуго лайбахи | Белая ржавчина | CCA27519 | предполагаемый белок, ассоциированный с вакуолярной мембраной | 1591 г. до н.э. | 20% | 46% | 1362 |
С момента расхождения животных и грибов сохранилось 30 остатков, 26 из которых расположены в домене DUF 3608. [16] Следующее множественное выравнивание последовательностей иллюстрирует эту консервативность домена DUF ; представители кладов беспозвоночных и грибов выровнены с человеческим DUF 3608, при этом полностью сохранившиеся остатки окрашены в зеленый цвет.

Паралоги
Не существует известных паралогов человеческого DEPDC5 , [14] но существует 64 человеческих белка, содержащих гомологичный домен DEP. [17] Также не выявлено паралогов для дрожжевого белка Iml1, наиболее отдаленного ортолога человеческого DEPDC5. [14]
Выражение
Экспрессия DEPDC5 была охарактеризована как повсеместная в тканях человека с помощью анализа ОТ-ПЦР [18] и исследований ДНК-микрочипов, как показано в таблице ниже. [19]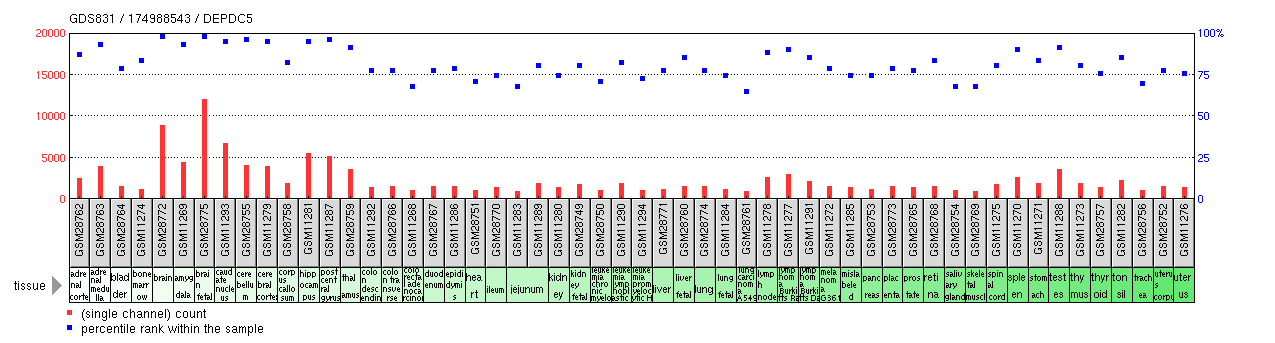
В одном исследовании пациентов с гепатоцеллюлярной карциномой была обнаружена более высокая экспрессия DEPDC5 в опухолевой ткани, чем в неопухолевой ткани. [5] Напротив, гомозиготная делеция трех генов, один из которых — DEPDC5, была обнаружена в двух случаях глиобластомы . [6] Другие аномалии экспрессии включают нулевую экспрессию в клеточной линии рака молочной железы MDA-MB-231 [20] и низкую экспрессию в клеточной линии P116 ( ZAP70- отрицательная). [21]
Посттрансляционные модификации
Следующие посттрансляционные модификации были предсказаны с помощью протеомных инструментов, составленных в ExPASy [22] и PhosphoSite Plus [23] для человеческого белка DEPDC5.
| Посттрансляционная модификация | Число/Местоположение | Источник |
|---|---|---|
| Фосфорилирование | 133/(Сер: 87 Трет: 23 Тир: 23) | NetPhos |
| 6/S579, S582, S1499, Y1515, Y1519, Y1543 | ФосфоСит Плюс | |
| Гликация | 29/5, 8, 13, 14, 28, 34, 56, 59, 64, 93, 131, 147, 229, 247, 256, 319, 436, 528, 609, 710, 862, 878, 1008, 1185, 1233, 1387, 1408, 1499, 1567, 1597 гг. | NetGlycate |
| Сайт N-гликозилирования | 9/Н201, Н298, Н311, Н384, Н684, Н1157, Н1377, Н1444, Н1529 | NetNGlyc |
| Сульфатирование | 3/Y397, Y459, Y462 | Сульфинатор |
| Сумоилирование | 2/К59, К147 | SUMOsp Архивировано 2013-05-10 в Wayback Machine |
| Расщепление пропептида | 2/R1004-M1005, R1528-N1529 | ProP |
| О-гликозилирование | 0 | NetOGlyc |
| C-маннозилирование | 0 | NetCGlyc |
| Миристоилирование | 0 | Миристоилирование |
| Пренилирование | 0 | PrePS Архивировано 2012-02-08 на Wayback Machine |
| Ацетилирование | 0 | NetAcet |
Взаимодействие
DEPDC5, возможно, может взаимодействовать с субъединицей протеасомы PSMA3 , о чем свидетельствует коиммунопреципитация [24] и фактор транскрипции MYC . [25] DEPDC5 находится в комплексе «GATOR1» с NPRL2 и NPRL3 . [26]
Ссылки
- ^ abc GRCh38: Ensembl выпуск 89: ENSG00000100150 – Ensembl , май 2017 г.
- ^ abc GRCm38: Ensembl выпуск 89: ENSMUSG00000037426 – Ensembl , май 2017 г.
- ^ "Human PubMed Reference:". Национальный центр биотехнологической информации, Национальная медицинская библиотека США .
- ^ "Mouse PubMed Reference:". Национальный центр биотехнологической информации, Национальная медицинская библиотека США .
- ^ abc Miki D, Ochi H, Hayes CN и др. (август 2011 г.). «Изменение в локусе DEPDC5 связано с прогрессированием гепатоцеллюлярной карциномы у носителей хронического вируса гепатита С». Nat. Genet . 43 (8): 797– 800. doi :10.1038/ng.876. PMID 21725309. S2CID 205357903.
- ^ ab Seng TJ, Ichimura K, Liu L, Tingby O, Pearson DM, Collins VP (июнь 2005 г.). «Сложные перестройки хромосомы 22 в астроцитарных опухолях, выявленные с помощью микросателлитного анализа и анализа массива путей плитки хромосомы 22». Гены Хромосомы Рак . 43 (2): 181– 93. doi :10.1002/gcc.20181. PMID 15770670. S2CID 45003453.
- ^ ab "GeneCards: Домен DEP, содержащий 5".
- ^ "AceView: сложный локус DEPDC5 Homo sapiens, кодирующий домен DEP, содержащий 5".
- ^ "ШВЕЙЦАРСКАЯ МОДЕЛЬ".
- ^ "NCBI: CBLAST".
- ^ Pan WJ, Pang SZ, Huang T, Guo HY, Wu D, Li L (август 2004 г.). «Характеристика функции трех доменов в dishevelled-1: домен DEP отвечает за мембранную транслокацию dishevelled-1». Cell Res . 14 (4): 324–30 . doi : 10.1038/sj.cr.7290232 . PMID 15353129.
- ^ «Рабочий стол биологии: PELE».
- ^ Wu X, Tu BP (ноябрь 2011 г.). «Избирательная регуляция аутофагии комплексом Iml1-Npr2-Npr3 при отсутствии азотного голодания». Mol. Biol. Cell . 22 (21): 4124– 33. doi :10.1091/mbc.E11-06-0525. PMC 3204073. PMID 21900499 .
- ^ abc "NCBI".
- ^ «TimeTree».
- ^ «Рабочий стол биологии: ClustalW».
- ^ Civera C, Simon B, Stier G, Sattler M, Macias MJ (февраль 2005 г.). «Структура и динамика домена DEP человеческого плекстрина: отличительные молекулярные особенности нового подсемейства доменов DEP». Proteins . 58 (2): 354– 66. doi :10.1002/prot.20320. PMID 15573383. S2CID 45722575.
- ^ Ishikawa K, Nagase T, Suyama M и др. (июнь 1998 г.). «Предсказание кодирующих последовательностей неидентифицированных генов человека. X. Полные последовательности 100 новых клонов кДНК из мозга, которые могут кодировать большие белки in vitro». DNA Res . 5 (3): 169–76 . doi : 10.1093/dnares/5.3.169 . PMID 9734811.
- ^ Джонсон Дж. М., Касл Дж., Гарретт-Энгеле П. и др. (декабрь 2003 г.). «Обзор генома альтернативного сплайсинга пре-мРНК человека с использованием микроматриц экзонных соединений». Science . 302 (5653): 2141– 4. Bibcode :2003Sci...302.2141J. doi :10.1126/science.1090100. PMID 14684825. S2CID 10007258.
- ^ Cappellen D, Schlange T, Bauer M, Maurer F, Hynes NE (январь 2007 г.). «Новые целевые гены c-MYC опосредуют дифференциальные эффекты на пролиферацию и миграцию клеток». EMBO Rep . 8 (1): 70– 6. doi :10.1038/sj.embor.7400849. PMC 1796762. PMID 17159920 .
- ^ Roose JP, Diehn M, Tomlinson MG и др. (ноябрь 2003 г.). «Т-клеточный рецептор-независимый базальный сигнал через киназы Erk и Abl подавляет экспрессию гена RAG». PLOS Biol . 1 (2): E53. doi : 10.1371 /journal.pbio.0000053 . PMC 261890. PMID 14624253.

- ^ "ExPASy Proteomic Tools: Post-translational modification predicting". Архивировано из оригинала 2012-04-24 . Получено 2012-05-08 .
- ^ «ФосфоСайт Плюс: DEPDC5».
- ^ Lim J, Hao T, Shaw C и др. (май 2006 г.). «Сеть взаимодействия белок-белок при наследственных атаксиях и расстройствах дегенерации клеток Пуркинье у человека». Cell . 125 (4): 801– 14. doi : 10.1016/j.cell.2006.03.032 . PMID 16713569.
- ^ Zeller KI, Zhao X, Lee CW и др. (ноябрь 2006 г.). «Глобальное картирование участков связывания c-Myc и сетей целевых генов в человеческих В-клетках». Proc. Natl. Acad. Sci. USA . 103 (47): 17834– 9. Bibcode : 2006PNAS..10317834Z. doi : 10.1073/pnas.0604129103 . PMC 1635161. PMID 17093053 .
- ^ Bar-Peled, L; Chantranupong, L; Cherniack, AD; Chen, WW; Ottina, KA; Grabiner, BC; Spear, ED; Carter, SL; Meyerson, M; Sabatini, DM (2013). "Комплекс супрессора опухолей с активностью GAP для Rag GTPases, которые сигнализируют о достаточности аминокислот для mTORC1". Science . 340 (6136): 1100– 6. Bibcode :2013Sci...340.1100B. doi :10.1126/science.1232044. PMC 3728654 . PMID 23723238.






