Геномика древних патогенов
Геномика древних патогенов — это научная область , связанная с изучением геномов патогенов, извлеченных из древних останков человека , растений или животных. [1] Древние патогены — это микроорганизмы , ныне вымершие, которые в прошлые столетия стали причиной нескольких эпидемий и смертей по всему миру. Их геном , называемый древней ДНК (аДНК), выделяют из останков захоронений (костей и зубов) жертв пандемий, вызванных этими патогенами.
Анализ геномных особенностей геномов древних патогенов позволяет исследователям понять эволюцию современных штаммов микроорганизмов, которые гипотетически могут генерировать новые пандемии или вспышки. Анализ ДНК проводится с помощью биоинформационных инструментов и методов молекулярной биологии для сравнения древних патогенов с современными потомками. Сравнение также предоставляет филогенетическую информацию об этих штаммах. [2]
Реконструкция геномов древних патогенов с помощью технологий NGS
Обнаружение патогенной ДНК в древних останках может быть достигнуто с помощью лабораторных или вычислительных методов. В обоих случаях процедура начинается с извлечения ДНК из древних образцов. Лабораторные методы основаны на построении библиотек NGS и последующем скрининге на основе захвата. Вычислительные инструменты используются для сопоставления прочтений, полученных с помощью NGS, с одно- или многогеномным эталоном (целевой подход); в качестве альтернативы могут применяться методы метагеномного профилирования или таксономического назначения дробовика прочтений NGS (широкий подход). [1]
Выделение древней ДНК
Ограниченная сохранность и, следовательно, низкая распространенность, сильно фрагментированное и поврежденное состояние, а также наличие современных загрязнений ДНК и фоновой ДНК из окружающей среды делают извлечение древней ДНК ( аДНК ) сложной процедурой. [ необходима ссылка ]
Для эффективного восстановления ДНК ДНК обычно изолируют из тканей, содержащих большое количество ДНК, таких как кости и зубы, которые в изобилии встречаются в археологических записях. [1] Сохранение патогенов в различных анатомических элементах очень варьируется в зависимости от типа патогена и его тканевого тропизма, пути проникновения в организм и вызванного им заболевания. Патогены, вызывающие хронические инфекции у своих хозяев, обычно вызывают диагностические изменения костей в отличие от острых инфекций, передающихся через кровь. [1] Поэтому для инфекций, которые привели к смерти хозяина в острой фазе, предпочтительным материалом для отбора проб является внутренняя полость зубов [1] [3], поскольку эта ткань сильно васкуляризирована в течение жизни. [4]
aDNA характеризуется повреждениями, которые накапливаются с течением времени: оценка «паттерна повреждений» ДНК с помощью вычислительных инструментов полезна для аутентификации древней патогенной ДНК, поскольку тот же самый паттерн не обнаруживается в современных загрязнителях. [5]
Наиболее представленное химическое повреждение, которое влияет на ДНК после смерти, — это гидролитическое дезаминирование цитозинов, превращающее их в урацилы, которые затем считываются как тимины. Из-за этой реакции древняя ДНК содержит неожиданную пропорцию переходов цитозина в тимин , в частности на концах молекул. [6] Другие распространенные модификации ДНК, помимо дезаминирования цитозина в тимин (это происходит, когда цитозины были метилированы), — это наличие абазических участков и одноцепочечных разрывов. [5]
ДНК сильно фрагментирована (большинство фрагментов имеют длину менее 100 пар оснований): эта тенденция может быть использована в качестве количественной меры подлинности, поскольку ожидается, что современные молекулы загрязняющих веществ будут длиннее. [4] Для использования этой характерной особенности древней ДНК были введены улучшенные протоколы экстракции на основе кремния с измененным объемом и составом буфера связывания ДНК . [5]
Создание библиотек ДНК
Для секвенирования с помощью методов секвенирования второго поколения необходимо модифицировать молекулы-шаблоны путем лигирования адаптеров. [7] Как этапы построения библиотеки, так и последующая ПЦР- амплификация подвержены ошибкам. В частности, могут возникнуть смещения в связывании адаптеров, а относительная эффективность ферментов ПЦР при амплификации конструкции может быть переменной. [5]
Существует три наиболее распространенных типа библиотек ДНК . Библиотека двухцепочечной ДНК использует шаблоны двухцепочечной ДНК и сначала требует шага для восстановления концов фрагментов ДНК. Затем фрагменты лигируются с двухцепочечными адаптерами, и полученные в результате разрывы заполняются . Этот метод имеет некоторые ограничения, такие как наличие фракции конструкций, которые не содержат оба различных адаптера, и возможное образование димеров адаптеров. [5]
Для преодоления этой последней проблемы был разработан метод построения библиотеки с А-хвостом. В этом методе aDNA подвергается репарации концов, а затем к 3'-концам нитей добавляется остаток аденина, что может облегчить лигирование шаблона с адаптерами, содержащими портной тимина. Кроме того, использование этих адаптеров с Т-хвостом предотвращает образование димеров адаптеров. Тип адаптера, который обычно используется, является двухцепочечным и имеет форму Y, что означает, что у него есть область, расположенная на конце с Т-хвостом, где он комплементарен, и область на другом конце, где он некомплементарен. Использование этого типа адаптеров позволяет генерировать шаблон aDNA, фланкированный различными некомплементарными последовательностями адаптеров на каждом конце, которые полезны для однонаправленного секвенирования. [5]
Другая стратегия основана на использовании библиотек одноцепочечной ДНК. В этом методе ДНК сначала денатурируется для получения одноцепочечной ДНК с помощью нагревания, а затем лигируется с одноцепочечным биотинилированным адаптером. Затем ДНК-цепь используется в качестве шаблона ДНК-полимеразой , которая производит комплементарную цепь. Затем второй адаптер лигируется на 3'-конце комплементарной цепи, и полная конструкция амплифицируется с помощью ПЦР, а затем секвенируется. Этап очистки выполняется с использованием покрытых стрептавидином парамагнитных шариков, которые позволяют минимизировать потерю ДНК на этом этапе процедуры. [5]
Обогащение библиотек для ДНК
Различные методы (называемые методами обогащения) были разработаны для улучшения доступности эндогенной ДНК в древних останках. Эти подходы можно в основном разделить на три типа: те, которые используются во время создания библиотеки, путем предпочтительного включения фрагментов ДНК, характеризующихся высоким уровнем повреждения, те, которые применяются после создания библиотеки, путем разделения экзогенных и эндогенных фракций посредством отжига с заранее определенными наборами зондов (в растворе или на микрочипах ), или те, которые основаны на целевом переваривании экологической микробной ДНК с использованием рестрикционных ферментов и захвата удлинения праймера (PEC). [5]
Селективное обогащение урацилом
Во время построения библиотеки фрагменты одноцепочечной ДНК связываются через биотинилированный адаптер с покрытыми стрептавидином бусинами. На этапе полимеразного удлинения генерируется цепь ДНК, комплементарная исходному шаблону. При таком обогащении конструкции подвергаются фосфорилированию на 5'-конце, чтобы обеспечить лигирование нефосфорилированного адаптера (лигирование между 3'-концом адаптера и 5'-концом вновь синтезированной цепи). Затем ДНК обрабатывается урацил-ДНК-гликозилазой (UDG) и эндонуклеазой VIII (смесь USER): UDG генерирует абазические сайты на цитозине, которые были дезаминированы в урацилы post-mortem, эндо VIII разрезает на полученном абазическом сайте. Это расщепление генерирует новые 3'-концы, которые затем дефосфорилируются, в результате чего образуются 3'OH-концы, которые можно использовать в качестве отправных точек для нового этапа удлинения. Это приводит к удлинению поврежденной нити от поврежденной области к ограниченной бусине: пока синтезируется новая молекула ДНК, исходный фрагмент смещается. В результате вновь образованные молекулы dsDNA больше не содержат адаптер, связанный с бусинами, оставляя в супернатанте библиотеку dsDNA нитей, которые изначально содержали дезаминированные цитозины, доступную для дальнейшей амплификации и секвенирования. Неповрежденная фракция шаблона ДНК остается прикрепленной к парамагнитным бусинам. [8]
Целевое обогащение без расширения в растворе
Этот подход основан на гибридизации мишени-зонда в растворе для скрининга только одного микроорганизма после создания библиотеки. Это видоспецифичный анализ, который требует тепловой денатурации библиотек ДНК и создания библиотеки зондов ДНК с использованием ПЦР дальнего действия, если доступен свежий материал ДНК близкородственных видов, или посредством индивидуального дизайна и синтеза олигонуклеотидов . [5] Этот метод полезен, когда известен целевой микроорганизм, например, когда существует гипотеза о возбудителе эпидемии или при наличии поражений скелета у изучаемых лиц. [1]
Твердофазное целевое обогащение
Другой обогащающей стратегией, применяемой после построения библиотеки, является прямое применение микрочипов . Они применяются для широкого лабораторного скрининга патогенов, который одновременно ищет различные патогенные микроорганизмы. Такой подход благоприятен для тех патогенов, которые не оставляют физических скелетных доказательств и чье присутствие не может быть легко предположено априори . Зонды разработаны для представления консервативных или уникальных областей из ряда патогенных вирусов, паразитов или бактерий. [5]
Поскольку микрочипы содержат последовательности, полученные из современных штаммов древних патогенов, ограничениями этого метода являются плохое обнаружение наиболее расходящихся геномных регионов и пропуск регионов с важными геномными перестройками или неизвестными дополнительными плазмидами . [5]
Обогащение всего генома
Захват всего генома в растворе (WISC) позволяет охарактеризовать всю последовательность генома древних людей. Этот метод основан на использовании библиотеки биотинилированных РНК-зондов по всему геному, созданной путем транскрипции in vitro свежих современных экстрактов ДНК из видов, тесно связанных с целевым образцом ДНК. Затем библиотека ДНК, денатурированная нагреванием, отжигается с зондами РНК. Для повышения строгости и снижения обогащения для высокоповторяющихся областей добавляются олигонуклеотиды ДНК низкой сложности и РНК-блокаторы адаптера. Затем интересующая фракция библиотеки восстанавливается путем элюирования из покрытых стрептавидином парамагнитных шариков (с которыми связаны зонды РНК). [9]
Вычислительный анализ
Анализ данных последовательностей, полученных с помощью NGS, опирается на те же вычислительные подходы, которые используются для современной ДНК, с некоторыми особенностями. Широко используемым инструментом для выравнивания прочтений из aDNA с референтными геномами является пакет PALEOMIX, который может количественно оценить уровни повреждений ДНК с помощью mapDamage2 и выполнить филогеномный и метагеномный анализы. Важно учитывать, что выравнивание всегда будет демонстрировать существенные доли несовпадающих нуклеотидов, которые являются результатом не ошибок секвенирования или полиморфизмов, а наличия поврежденных оснований. По этой причине порог приемлемости для расстояния редактирования от чтения до референта должен быть выбран в соответствии с филогенетическим расстоянием до референтного генома. Для улучшения выравниваний были разработаны вероятностные выравниватели, которые учитывают характер повреждений aDNA. [5]
СОЛОД
Исследования древней ДНК патогенов ограничены скелетными коллекциями, которые меняют свой внешний вид в результате инфекций. Патоген, связанный с известным эпидемиологическим контекстом, идентифицируется посредством скрининга без предварительного знания о его присутствии. Методы включают молекулярные подходы широкого спектра, ориентированные на обнаружение патогена с помощью технологии микрочипов на основе флуоресцентной гибридизации, идентификацию с помощью обогащения ДНК определенных микробных регионов или вычислительный скрининг необогащенных данных о последовательностях против наборов данных человеческого микробиома . Эти подходы предлагают улучшения, но остаются предвзятыми в бактериальных таксонах, используемых для назначений на уровне видов. [10]
Инструмент выравнивания MEGAN (MALT) — это новая программа для быстрого метода выравнивания и таксономического назначения для идентификации древней ДНК. MALT похож на BLAST , поскольку он вычисляет локальные выравнивания между высококонсервативными последовательностями и ссылками. MALT также может вычислять полуглобальные выравнивания, где чтения выравниваются от начала до конца. Все ссылки, полные бактериальные геномы, содержатся в базе данных под названием RefSeq Национального центра биотехнологической информации (NCBI). MALT состоит из двух программ: malt-build и malt-run. Malt-build используется для построения индекса для заданной базы данных референтных последовательностей. Вместо этого malt-run используется для выравнивания набора последовательностей запроса с референтной базой данных. Затем программа вычисляет бит-счет и ожидаемое значение (E-значение) выравнивания и решает, сохранить или отбросить выравнивание в зависимости от указанных пользователем пороговых значений для бит-счета, E-значения или процента идентичности. Бит-счет — это требуемый размер базы данных последовательностей, в которой текущее совпадение может быть найдено просто случайно. Чем выше бит-счет, тем лучше сходство последовательностей. E-значение — это количество ожидаемых совпадений похожего качества (счет), которые могут быть найдены просто случайно. Чем меньше E-значение, тем лучше совпадение. [10]
MALT позволяет проводить скрининг необогащенных данных о последовательностях при поиске неизвестных кандидатов на бактериальные патогены, которые были вовлечены в прошлые вспышки заболеваний, и для исключения бактериального фона окружающей среды. MALT очень важен, поскольку он дает преимущество скрининга на уровне генома без выбора конкретного целевого организма, избегая ошибок, которые являются общими для других подходов к скринингу. Для аутентификации кандидатов на таксономические назначения необходимы полные выравнивания, но целевая ДНК часто присутствует в небольшом количестве, поэтому небольшого количества отмеченной области может быть недостаточно для идентификации. Этот подход позволяет обнаружить только бактериальную ДНК и вирусную ДНК, поэтому невозможно идентифицировать других инфекционных агентов, которые могут присутствовать в популяции. Этот метод полезен для исследований, связанных с идентификацией патогенов, ответственных за древние и современные заболевания, особенно в случаях, когда кандидаты на организмы неизвестны априори . [10]
Приложения
Геномика древних патогенов как инструмент против будущих эпидемий
Одним из интересных применений различных методов секвенирования, доступных в настоящее время, является исследование исторических вспышек заболеваний с целью получения ответа на важные и давние вопросы в эпидемиологии, эволюции патогенов, а также истории человечества. [2]
Итак, много усилий тратится на то, чтобы найти все больше и больше информации об этиологии инфекционных заболеваний, имеющих историческое значение, таких как чума и эпидемия коколизтли , чтобы описать географическое распространение вирусов и попытаться определить патогенный механизм этих инфекционных агентов, которые на самом деле являются активными элементами эволюционного процесса. Сегодня Y.pestis и S. enterica кажутся безвредными для человека, но ученые по-прежнему заинтересованы в долгосрочном отслеживании генетической адаптации этих бактерий и точной количественной оценке темпов их эволюционных изменений. Это потому, что они могут извлечь из этих знаний прошлого правильные идеи для разработки стратегии против будущих эпидемий. [2]
Прекрасно осознавая тот факт, что бактерии и вирусы являются одними из самых изменчивых элементов в природе, подверженными неограниченным мутационным событиям, и принимая как должное, что невозможно управлять всеми внешними факторами, которые могут повлиять на развитие патогенного вируса, никто не говорит о победе над новой возможной вспышкой чумы или любого другого инфекционного агента прошлого: здесь цель состоит в том, чтобы определить стратегию, «руководство», чтобы быть более подготовленными, когда появится новый опасный патоген. Вклад окружающей среды в инфекции должен быть определен, и такие факторы, как миграция людей, изменение климата, перенаселенность городов или одомашнивание животных, являются одними из основных причин, которые способствуют возникновению и распространению болезней. Конечно, эти факторы непредсказуемы, и это причина, по которой исследователи пытаются извлечь из прошлого соответствующую информацию, которая может быть полезна сегодня и завтра. Продолжая разрабатывать стратегии для победы над возникающими угрозами с использованием диагностических, молекулярных и передовых инструментов, они все еще оглядываются на то, как древние патогены развивались и адаптировались в ходе исторических событий. Чем больше известно о геномной основе вирулентности исторических заболеваний, тем больше можно понять о возникновении и повторном появлении инфекционных заболеваний сегодня и в будущем. [2]
Древние инфекции и эволюция человека
Анализ филогенетических отношений между человеком-хозяином и вирусными патогенами показывает, что многие заболевания развивались совместно с людьми на протяжении тысячелетий, с самого начала человеческой истории в Африке. [ необходима цитата ]
В частности, долгосрочное взаимодействие с патогенами считается отбором, который может быть очень сильным, поскольку не все особи могли выжить при контакте со всеми инфекционными агентами, с которыми они встречались на протяжении многих лет: естественный отбор патогенами участвует в эволюции видов. Это взаимодействие уже использовалось для отслеживания перемещений населения и реконструкции потоков миграции людей в пределах и за пределами Африки. [11]
Довольно новое применение и интерпретация этой особенности — использование ДНК для лучшего понимания эволюции человека. Многие тропические инфекции, вероятно, сыграли значительную роль в процессе эволюции человека. Корреляция между людьми и вирусами может быть понята, если рассматривать ее как «борьбу», которая продолжается тысячелетиями и в которой до сих пор никто не победил: когда вирусы изменили свои характеристики, чтобы стать заразными для других «бойцов», людям пришлось найти стратегию для повышения своей приспособленности и выжить среди изменений. [11]
В этом непрерывном вызове на протяжении многих лет, рядом с инфекционными заболеваниями и другими болезнями, поражающими современное человеческое общество, рак в последнее время представляет собой одно из самых загадочных заболеваний. Ученые исследуют, ограничены ли неопластические заболевания постиндустриальным человеческим обществом или их истоки можно найти еще дальше во времени, может быть, в доисторические времена. Трудность заключается в том, что рак, смертельный и быстрый, оставляет очень мало следов в скелетах в тех случаях, когда смерть наступает быстро, и даже не оставляет никаких признаков существования вообще в случае внескелетных опухолей. В любом случае, знания об этиологии рака неполны, и микроорганизмы берут на себя роль своей инфекции: миграционные перемещения в прошлом могли принести с собой вирусы, поэтому возможный резервуар тропических болезней, а также предрасположенность к раку. По этой причине молекулярные аналитические методы применяются к археологическим останкам для изучения эволюции гомининов, а также для улучшения исследований в понимании эпидемиологии и этиологии опухолей. Информация, полученная из ДНК, может быть использована для фиксации мутаций патогенов и реконструкции эволюционного процесса на основе присутствия микроорганизмов. Она может быть полезна для разработки новых вакцин или для обнаружения возможных будущих патогенных угроз. [11]
Прошлые пандемии — это нечто большее, чем просто древняя история
То, что произошло в прошлом, не является историей, есть что-то скрытое, что все еще может управлять генетическим разнообразием человека и естественным отбором, что-то, что вошло в контакт с человечеством сотни лет назад, но что все еще может оказывать влияние на глобальное здоровье человека. Поскольку эпидемии являются одним из наиболее частых явлений, которые затронули и потенциально опустошили человеческие популяции, важно обнаруживать, предотвращать и контролировать потенциальных инфекционных агентов. В конце концов, археологи, генетики и ученые-медики занимаются изучением влияния патогенов, которые могут способствовать, угрожать или улучшать здоровье и долголетие человека. [11]
Эволюция и филогенезYersinia pestis
Yersinia pestis — грамотрицательная бактерия, относится к семейству Enterobatteriaceae. Ближайшими ее родственниками являются Yersinia pseudotuberculosis и Yersinia enterocolitica , которые являются экологическими видами. [ необходима цитата ]
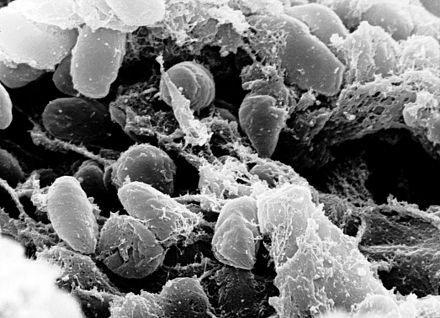
Все они обладают плазмидой pCD1, которая кодирует секреторную систему типа III. Среди генов, кодирующих хромосомные белки, их нуклеотидная геномная идентичность составляет 97%. Они различаются по своему потенциалу вирулентности и механизмам передачи. [12]
Y. pestis не является бактерией, адаптированной к человеку. Ее основными резервуарами являются грызуны (такие как сурки, мыши, большие песчанки, полевки и луговые собачки), а передается она человеку через блох . Одним из наиболее изученных векторов этого патогена является Xenopsylla cheopis . [ необходима цитата ]
После укуса инфицированной блохи бактерии проникают в организм хозяина и перемещаются в ближайший лимфатический узел, где бактерии размножаются, вызывая большие опухоли, называемые бубонами. Бактерии также могут распространяться в кровоток (вызывая септицемию) и в легкие (вызывая пневмонию). Легочное заболевание имеет прямой путь передачи от человека к человеку. [13]
Было установлено, что Y. pestis стал таким опасным из-за приобретения ymt (мышиный токсин йерсины). Этот ген присутствует в плазмиде pMT1 и позволил бактерии выжить в переносчике-блохе и способствовал колонизации средней кишки членистоногих, что привело к крупномасштабным пандемиям прошлого тысячелетия. [13]
Ранняя эволюция и расхождение отYersinia pseudotuberculosis
Y. pestis отличается от двух других видов своей патогенностью и механизмом передачи. Эти различия обусловлены двумя плазмидами: pPCP1, которая придает бактерии ее инвазивные свойства в отношении людей, и pMT1, которая участвует в колонизации блох (наряду с некоторой потерей функции на бактериальных хромосомных генах). [ необходима цитата ]
Образцы, датированные поздним неолитом и бронзовым веком, позволили идентифицировать первую генетическую дивергенцию между предками Y. pseudotuberculosis и Y. pestis . Характеристики, которые придают Y. pestis его вирулентность, отсутствовали в этих штаммах: у них отсутствует ymt , ген, необходимый для колонизации вектора; также они представляли активную форму генов, необходимых для образования биопленки (неактивных у патогена Y. pestis ) и активный ген флагеллина , который является индуктором иммунного ответа (является псевдогеном у Y. pestis ). [1]
Сравнение проекта генома и двух плазмид (pCD1 и pMT1) с образцами жертв Черной смерти (1348–1349) в могильнике Восточный Смитфилд подчеркнуло очень высокую генетическую консервативность последовательности: всего 97 однонуклеотидных различий за 660 лет. [14]
Y. чумная палочкамикроэволюция
Отдельный штамм London 6330 обладает мутациями, отсутствующими в других изолятах того же периода (1348–1350 гг.): причиной может быть либо наличие нескольких штаммов, циркулирующих в Европе в одно и то же время, либо микроэволюция одного штамма во время пандемии. [14]
Три крупные вспышки чумы
Существует три пандемические вспышки Y. Pestis : [ необходима ссылка ]
- Первая известна как чума Юстиниана, она впервые случилась в Египте в 541-543 годах, а затем распространилась на Константинополь и соседние регионы. Вспышки в Европе наблюдались до 750 года н. э. Филогенетический анализ показал, что оба генома принадлежат к линии, которая сегодня вымерла и тесно связана со штаммами из современного Китая, что предполагает возможность восточноазиатского происхождения первой пандемии.
- Вторая пандемия известна как Черная смерть или Великая чума. Она произошла в 1346–1352 годах в Европе и сопровождалась многочисленными вспышками чумы, она продолжалась до 18 века. Возможно, что в этой пандемии было два разных штамма Y. pestis , которые проникли на континент через разные импульсы.
- Третья пандемия началась в Китае в 1860 году. Она быстро распространилась на другие страны из-за использования железных дорог и пароходов.
Штаммы, связанные с Юстиниановой чумой, появляются на новой ветви, которая филогенетически отличается от второй и третьей пандемий чумы. Первый штамм Y. pestis, обнаруженный во время второй вспышки, выживает и дает начало современным штаммам ветви 1, связанным с третьей вспышкой пандемии. [ необходима цитата ]
Первая бактерия чумы, а также второй и третий штаммы чумы имеют общего предка. [2]
Связь между 2-й и 3-й пандемиями
В недавнем исследовании [15] геномы Y. pestis из трех образцов были возобновлены в Барселоне (умер между 1300 и 1420 годами), Эльвангене (между 1486 и 1627 годами) и городе Болгар (между 1298 и 1388 годами). Дата смерти проанализированных лиц была определена благодаря радиоуглеродному датированию ; последнее было подтверждено наличием монеты, произведенной только после 1362 года. Из 223 образцов от 178 лиц только один для каждого места имел подходящее количество ДНК и был окончательно выбран для полного геномного секвенирования бациллы (с помощью анализа захвата генома, используя в качестве черновика геном Y. pseudotuberculosis и pMT1 и pCD1 из Y. pestis ). [ необходима цитата ]
Выравнивание с филогенетическим деревом Y. pestis, созданным с использованием ранее известных древних геномов, выявило увеличение генетического разнообразия за пределами Китая по сравнению с тем, что считалось ранее; все три новых генома, отображенные в ветви 1, обладают двумя SNP , связанными с Черной смертью (все геномы Y. pestis датированы картой Черной смерти в ветви 1). [14] Штамм Barcelona не имеет различий со штаммом London; два человека, от которых был получен геном, умерли от чумы с разницей в несколько месяцев (весной и осенью 1348 года [16] [17] ), что подчеркивает присутствие в Европе одной волны чумы с низким генетическим разнообразием. Штамм Ellwangen отображается в подветви ветви 1 и является предком ранее секвенированного штамма (L'Observance). [18] Он происходит от штамма, циркулировавшего в Лондоне и Барселоне во время Черной смерти, но также имеет дополнительные мутации. Поэтому считается, что эта линия отделилась от ветви 1 до 16-го века (вспышка в Эльвангене) и не имеет известных современных потомков. [15]
По сравнению с изолятами от Черной смерти штамм из города Болгар демонстрирует:
- p3 и p4, общие для «лондонского индивидуума 6330»;
- p6, общий для всех современных штаммов Branch 1;
- p7, уникальный для этого штамма;
Штамм из Болгар-Сити обладает однонуклеотидными полиморфизмами, связанными с Черной смертью, и может быть свидетельством перемещения чумы на восток. Эти результаты подтверждают одну из моделей, пытающихся объяснить связь между 2-й и 3-й пандемией: в этом сценарии был один выход чумы в Европу (вызвавший Черную смерть), которая после радиационного события переместилась на восток, чтобы обосноваться в бывшем Советском Союзе и Азии, откуда она распространилась в 18 веке, вызвав 3-ю пандемию. [15]
Другая гипотеза заключается в том, что линия 3-й пандемии могла быть создана ранее существовавшей генетической изменчивостью штаммов Y. pestis в Китае: эта гипотеза фактически подтверждается корреляцией между последующими волнами пандемии в Европе и климатическими колебаниями, которые могли бы позволить ей распространиться на континенте. Эта модель не может объяснить генетическое разнообразие Черной смерти (по крайней мере, четыре различных линии, которые потребовали бы завоза из Азии четырех различных штаммов). [19]
Опять же, есть две модели, которые пытаются объяснить многочисленные вспышки чумы в Европе после «черной смерти»:
- Повторное занесение чумы из Азии. Этот сценарий совместим со 2-й теорией, обсуждавшейся ранее, которая рассматривает генетическую изменчивость Y. pestis в Китае; [20]
- Наличие в Европе резервуара ( ныне вымершего), вызывавшего постоянные вспышки вплоть до XVIII века;
Обе модели могут быть действительными, и в настоящее время мы не можем продемонстрировать одну над другой. Однако геном штамма Ellwangen, секвенированный в этом исследовании, можно считать доказательством второй гипотезы из-за географического положения города, которое, как правило, исключает возможность заноса чумы с востока. [15]
СовременныйY. чумная палочкаштаммы
Секвенирование геномов Y. pestis позволило обнаружить событие, предшествовавшее Черной смерти, которое дало начало многим штаммам, циркулирующим сегодня. [15]
Сальмонелла энтерикаанализ геномов
Серия 16-х эпидемий в Мексике, называемая « huey cocoliztli » на языке коренных народов науатль, вызвала высокую смертность среди коренного населения ацтеков , что привело к демографическому коллапсу. [21] Эти эпидемии считаются одними из самых страшных эпидемий в истории Мексики, а их причины остаются загадкой на протяжении более 500 лет. [ требуется ссылка ]
Группа ученых из Гарварда и Института Макса Планка опубликовала исследование в журнале Nature ecology and Evolution , в котором они предлагают Salmonella enterica в качестве хорошего кандидата для сильной эпидемии в Мексике в 16 веке. [10] Многие исследования предполагают, что эта бактерия была завезена в коренное население европейцами. [ необходима ссылка ]
Группа ученых проанализировала ДНК, извлеченную из зубов 24 скелетов, захороненных на кладбище в городе Тепосколула-Юкундаа, и обнаружила в 10 из 24 скелетов следы ДНК Salmonella enterica . Кроме того, чтобы продемонстрировать, что бактерия была завезена в Мексику европейцами, они проанализировали пять человек, которые были захоронены до притока европейцев. Результаты показали, что не было никаких доказательств наличия Salmonella enterica в доконтактную эпоху . [10]
АнализСальмонелла энтерикагеномы
Ученые провели извлечение ДНК из зубов 24 коренных жителей с эпидемического кладбища контактной эпохи и 5 человек, похороненных на кладбище доконтактной эпохи. Извлечение было проведено в соответствии с протоколом для извлечения ДНК. Группа исследователей параллельно также исследовала образцы почвы кладбищ, чтобы получить обзор микроорганизмов окружающей среды, которые могли проникнуть в образцы. [ необходима цитата ]
После извлечения геномы были секвенированы с помощью геномного анализатора Illumina. Затем, используя биоинформатический инструмент MALT, исследователи провели анализ данных метагеномных последовательностей. Эта программа позволяет исследователям выравнивать извлеченные последовательности с эталоном без указания точной цели. Исследователи выполнили MALT-прогон два раза: один раз с использованием полных бактериальных геномов, которые были доступны через NCBI (Национальный центр биотехнологической информации) RefSeq в качестве эталона, а второй прогон был выполнен с использованием полной базы данных NCBI Nucleotide для скрининга вирусной ДНК. [ необходима цитата ]
Результат процесса скрининга был положительным на наличие ДНК Salmonella enterica в 10 последовательностях до 24, собранных из образцов, и три образца зубов имели большое количество прочтений, отнесенных к S. enterica . В частности, основным штаммом S. enterica , присутствующим в образцах, является S. Paratyphi C. Этот штамм вызывает брюшной тиф у людей. В образцах доконтактной эпохи они не нашли никаких доказательств наличия S. enterica , что подтверждает гипотезу о том, что S. enterica не была местной бактерией. [ необходима цитата ]
Дальнейший анализ был проведен для выявления классической модели повреждения аДНК в трех положительных образцах зубов, и это было сделано путем сопоставления наборов данных с эталонным геномом S. Paratyphy C. Результаты были положительными и подтвердили тезис о S.enterica как причине коколицли.
Чтобы углубить анализы и подтвердить тезис, исследователи провели дополнительные эксперименты и вычислительный анализ. Они выполнили целевую матрицу всего генома и гибридизацию в растворе, используя зонды, которые включают современные различия генома S. enterica и используя S. Paratyphi C в качестве референса. Гибридизация прошла успешно для десяти положительных образцов, в то время как другие образцы дали отрицательный результат для древней ДНК. [10]
Смотрите также
Ссылки
- ^ abcdefg Spyrou, Maria A.; Bos, Kirsten I.; Herbig, Alexander; Krause, Johannes (2019-04-05). «Геномика древних патогенов как новый инструмент для исследования инфекционных заболеваний». Nature Reviews Genetics . 20 (6): 323– 340. doi :10.1038/s41576-019-0119-1. ISSN 1471-0056. PMC 7097038. PMID 30953039 .
- ^ abcde Andam, Cheryl P.; Worby, Colin J.; Chang, Qiuzhi; Campana, Michael G. (декабрь 2016 г.). «Микробная геномика древних эпидемий и вспышек». Trends in Microbiology . 24 (12): 978– 990. doi :10.1016/j.tim.2016.08.004. ISSN 1878-4380. PMID 27618404.
- ^ Маргарян, Ашот; Хансен, Генрик Б.; Расмуссен, Саймон; Сикора, Мартин; Моисеев, Вячеслав; Хохлов, Александр; Эпимахов, Андрей; Епископосян, Левон; Крииска, Айвар; Варул, Лииви; Сааг, Лехти (2018-02-26). "ДНК древних патогенов в человеческих зубах и каменистых костях". Экология и эволюция . 8 (6): 3534– 3542. Bibcode : 2018EcoEv...8.3534M. doi : 10.1002/ece3.3924 . ISSN 2045-7758. PMC 5869295. PMID 29607044 .
- ^ ab Key, Felix M.; Posth, Cosimo; Krause, Johannes; Herbig, Alexander; Bos, Kirsten I. (август 2017 г.). «Mining Metagenomic Data Sets for Ancient DNA: Recommended Protocols for Authentication». Trends in Genetics . 33 (8): 508– 520. doi : 10.1016/j.tig.2017.05.005. ISSN 0168-9525. PMID 28688671.
- ^ abcdefghijkl Орландо, Людовик; Гилберт, М. Томас П.; Виллерслев, Эске (2015-06-09). «Реконструкция древних геномов и эпигеномов». Nature Reviews Genetics . 16 (7): 395– 408. doi :10.1038/nrg3935. ISSN 1471-0056. PMID 26055157. S2CID 8440755.
- ^ Gilbert, MTP; Binladen, J.; Miller, W.; Wiuf, C.; Willerslev, E.; Poinar, H.; Carlson, JE; Leebens-Mack, JH; Schuster, SC (2006-12-07). "Перехарактеризация древних повреждений ДНК, связанных с неправильным кодированием: идеи в эпоху секвенирования через синтез". Nucleic Acids Research . 35 (1): 1– 10. doi : 10.1093/nar/gkl483 . ISSN 0305-1048. PMC 1802572. PMID 16920744 .
- ^ Гудвин, Сара; Макферсон, Джон Д.; МакКомби, У. Ричард (17.05.2016). «Совершеннолетие: десять лет технологий секвенирования следующего поколения». Nature Reviews Genetics . 17 (6): 333– 351. doi :10.1038/nrg.2016.49. ISSN 1471-0056. PMC 10373632. PMID 27184599. S2CID 8295541 .
- ^ Gansauge, Marie-Theres; Meyer, Matthias (сентябрь 2014 г.). «Избирательное обогащение поврежденных молекул ДНК для секвенирования древнего генома». Genome Research . 24 (9): 1543– 1549. doi :10.1101/gr.174201.114. ISSN 1088-9051. PMC 4158764 . PMID 25081630.
- ^ Карпентер, Мередит Л. Буэнростро, Джейсон Д. Вальдиосера Моралес, Кристина Шредер, Ханнес Аллентофт, Мортен Эрик Сикора, Мартин Расмуссен, Мортен Гравел, Симон Гильен, Соня Нехризова, Георгий Лештаков, Красимир Димитрова, Диана Теодосьев, Никола Петтенер, Давиде Луизелли , Доната Сандовал, Карла Морено-Эстрада, Андрес Ли, Ингруй Ван, Джун Гилберт, Том Виллерслев, Эске Гринлиф, Уильям Дж. Бустаманте, Карлос Д. (07.11.2013). «Извлечение 1%: захват всего генома для целевого обогащения библиотек секвенирования древней ДНК». Американский журнал генетики человека . 93 (5): 852– 864. doi :10.1016/j.ajhg.2013.10.002. OCLC 1035205487. PMC 3824117. PMID 24568772 .
{{cite journal}}: CS1 maint: несколько имен: список авторов ( ссылка ) - ^ abcdef Vågene, Åshild J.; Herbig, Alexander; Campana, Michael G.; Robles García, Nelly M.; Warinner, Christina; Sabin, Susanna; Spyrou, Maria A.; Andrades Valtueña, Aida; Huson, Daniel; Tuross, Noreen; Bos, Kirsten I. (март 2018 г.). "Геномы Salmonella enterica у жертв крупной эпидемии шестнадцатого века в Мексике". Nature Ecology & Evolution . 2 (3): 520– 528. Bibcode : 2018NatEE...2..520V. doi : 10.1038/s41559-017-0446-6. ISSN 2397-334X. PMID 29335577. S2CID 3358440.
- ^ abcd Рифкин, Риан Ф.; Потгитер, Марни; Рамон, Жан-Батист; Коуэн, Дон А. (декабрь 2017 г.). «Древний онкогенез, инфекция и эволюция человека». Эволюционные приложения . 10 (10): 949–964 . Бибкод : 2017EvApp..10..949R. дои : 10.1111/eva.12497. ISSN 1752-4571. ПМЦ 5680625 . ПМИД 29151852.
- ^ Chain, PSG; Carniel, E.; Larimer, FW; Lamerdin, J.; Stoutland, PO; Regala, WM; Georgescu, AM; Vergez, LM; Land, ML; Motin, VL; Brubaker, RR (2004-09-21). "Взгляд на эволюцию Yersinia pestis посредством сравнения всего генома с Yersinia pseudotuberculosis". Труды Национальной академии наук Соединенных Штатов Америки . 101 (38): 13826– 13831. Bibcode : 2004PNAS..10113826C. doi : 10.1073/pnas.0404012101 . ISSN 0027-8424. PMC 518763 . PMID 15358858.
- ^ ab Hinnebusch, B. Joseph; Rudolph, Amy E.; Cherepanov, Peter; Dixon, Jack E.; Schwan, Tom G.; Forsberg, Ake (2002-04-26). "Роль токсина Yersinia murine в выживании Yersinia pestis в средней кишке переносчика блох". Science . 296 (5568): 733– 735. Bibcode :2002Sci...296..733H. doi :10.1126/science.1069972. ISSN 1095-9203. PMID 11976454. S2CID 34770234.
- ^ abc Бос, Кирстен И.; Шунеманн, Верена Дж.; Голдинг, Дж. Брайан; Бурбано, Эрнан А.; Ваглехнер, Николас; Кумбс, Брайан К.; Макфи, Джозеф Б.; ДеВитт, Шэрон Н .; Мейер, Матиас; Шмедес, Сара; Вуд, Джеймс (октябрь 2011 г.). «Проект генома Yersinia pestis жертв Черной смерти». Природа . 478 (7370): 506–510 . Бибкод : 2011Natur.478..506B. дои : 10.1038/nature10549. ISSN 1476-4687. ПМК 3690193 . ПМИД 21993626.
- ^ abcde Spyrou, Maria A.; Tukhbatova, Rezeda I.; Feldman, Michal; Drath, Joanna; Kacki, Sacha; Beltrán de Heredia, Julia; Arnold, Susanne; Sitdikov, Airat G.; Castex, Dominique; Wahl, Joachim; Gazimzyanov, Ilgizar R. (2016-06-08). "Исторические геномы Y. pestis раскрывают европейскую черную смерть как источник древних и современных пандемий чумы". Cell Host & Microbe . 19 (6): 874– 881. doi : 10.1016/j.chom.2016.05.012 . ISSN 1931-3128. PMID 27281573.
- ^ Готтфрид, Роберт Стивен (1983-01-01). Черная смерть: природные и человеческие катастрофы в средневековой Европе. Free Press. ISBN 978-0-02-912630-1.
- ^ Бенедиктов, Оле Йорген (2006). Черная смерть, 1346-1353: Полная история. Boydell Press. ISBN 978-1-84383-214-0.
- ^ Бос, Кирстен И.; Хербиг, Александр; Саль, Джейсон; Ваглехнер, Николас; Фоурмент, Матье; Форрест, Стивен А.; Кланк, Дженнифер; Шуенеманн, Верена Дж.; Пойнар, Деби; Куч, Мелани; Голдинг, Г. Брайан (2016-01-21). "Геномы Yersinia pestis восемнадцатого века раскрывают долгосрочное существование исторического очага чумы". eLife . 5 : e12994. doi : 10.7554/eLife.12994 . ISSN 2050-084X. PMC 4798955 . PMID 26795402.
- ^ Хэнш, Стефани; Бьянуччи, Раффаэлла; Сигнили, Мишель; Раджерисон, Миноаризоа; Шульц, Майкл; Кацки, Саша; Вермунт, Марко; Уэстон, Дарлин А.; Херст, Дерек; Ахтман, Марк; Карниэль, Элизабет (07.10.2010). «Отдельные клоны Yersinia pestis вызвали Черную смерть». PLOS Pathogens . 6 (10): e1001134. doi : 10.1371/journal.ppat.1001134 . ISSN 1553-7374. PMC 2951374. PMID 20949072 .
- ^ Шмид, Борис В.; Бюнтген, Ульф; Истердей, В. Райан; Гинзлер, Кристиан; Валлоэ, Ларс; Браманти, Барбара; Стенсет, Нильс Хр (10.03.2015). «Климатически обусловленное внедрение Черной смерти и последовательные повторные заносы чумы в Европу». Труды Национальной академии наук . 112 (10): 3020– 3025. Bibcode : 2015PNAS..112.3020S. doi : 10.1073/pnas.1412887112 . ISSN 0027-8424. PMC 4364181. PMID 25713390 .
- ^ Acuna-Soto, Rodolfo; Stahle, David W.; Cleaveland, Malcolm K.; Therrell, Matthew D. (апрель 2002 г.). «Мегазасуха и мегасмерть в Мексике XVI века». Emerging Infectious Diseases . 8 (4): 360–362 . doi :10.3201/eid0804.010175. ISSN 1080-6040. PMC 2730237. PMID 11971767 .