ИНАВА
| ИНАВА | |||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Идентификаторы | |||||||||||||||||||||||||||||||||||||||||||||||||||
| Псевдонимы | INAVA , открытая рамка считывания хромосомы 1 106, C1orf106, активатор врожденного иммунитета | ||||||||||||||||||||||||||||||||||||||||||||||||||
| Внешние идентификаторы | ОМИМ : 618051; МГИ : 1921579; Гомологен : 10103; GeneCards : ИНАВА; ОМА :ИНАВА - ортологи | ||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Викиданные | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
INAVA , иногда называемый гипотетическим белком LOC55765 , представляет собой белок с неизвестной функцией, который у людей кодируется геном INAVA . [5] Менее распространенные псевдонимы гена включают FLJ10901 и MGC125608.
Ген
Расположение
У людей INAVA находится на длинном плече хромосомы 1 в локусе 1q32.1. Он охватывает от 200 891 499 до 200 915 736 (24,238 кб) на плюс-цепи. [5]
Джин соседство

INAVA фланкирован рецептором 25, связанным с G-белком (выше по течению), и членом семейства повторов maestro heat-like 3 (MROH3P), предсказанным нижестоящим псевдогеном. Псевдоген 6 рибосомного белка L34 (RPL34P6) находится выше по течению, а член семейства кинезинов 21B — ниже по течению. [5]
Промоутер

Существует семь предсказанных промоторов для INAVA, и экспериментальные данные свидетельствуют о том, что изоформы 1 и 2, наиболее распространенные изоформы, транскрибируются с использованием разных промоторов. [6] MatInspector, инструмент, доступный через Genomatix, использовался для прогнозирования участков связывания факторов транскрипции в потенциальных областях промотора. Факторы транскрипции, которые, как прогнозируется, нацелены на ожидаемый промотор для изоформы 1, экспрессируются в ряде тканей. Наиболее распространенными тканями экспрессии являются мочеполовая система, нервная система и костный мозг. Это совпадает с данными по экспрессии для белка INAVA, который высоко экспрессируется в почках и костном мозге. [7] Справа показана схема предсказанной области промотора с выделенными участками связывания факторов транскрипции. Факторы, которые, как прогнозируется, связываются с областью промотора изоформы 2, различаются, и двенадцать из двадцати лучших предсказанных факторов экспрессируются в клетках крови и/или тканях сердечно-сосудистой системы.
Выражение
C1orf106 экспрессируется в широком спектре тканей. Данные по экспрессии из профилей GEO показаны ниже. Места наибольшей экспрессии перечислены в таблице. Экспрессия умеренная в плаценте, простате, яичках, легких, слюнных железах и дендритных клетках. Она низкая в мозге, большинстве иммунных клеток, надпочечниках, матке, сердце и адипоцитах. [7] Данные по экспрессии, полученные в ходе различных экспериментов, обнаруженных в профилях GEO, свидетельствуют о том, что экспрессия INAVA повышается при нескольких видах рака, включая: рак легких, яичников, колоректальный рак и рак молочной железы.

| Салфетка | Процентильный ранг |
|---|---|
| В-лимфоциты | 90 |
| Трахея | 89 |
| Кожа | 88 |
| Клетки эпителия бронхов человека | 88 |
| Колоректальная аденокарцинома | 87 |
| Почка | 87 |
| Язык | 85 |
| Поджелудочная железа | 84 |
| Приложение | 82 |
| Костный мозг | 80 |
мРНК
Изоформы
Ген INAVA производит девять предполагаемых изоформ, семь из которых, как предполагается, кодируют белки. [8] Изоформы 1 и 2, показанные ниже, являются наиболее распространенными изоформами.

Изоформа 1, которая является самой длинной, принята в качестве канонической изоформы. Она содержит десять экзонов, которые кодируют белок длиной 677 аминокислот, в зависимости от источника. Некоторые источники сообщают, что белок состоит всего из 663 аминокислот из-за использования стартового кодона, который находится на сорок два нуклеотида ниже по течению. Согласно NCBI, эта изоформа была предсказана только вычислительным путем. [5] Это может быть связано с тем, что последовательность Козака , окружающая стартовый кодон ниже по течению, больше похожа на консенсусную последовательность Козака, как показано в таблице ниже. Softberry был использован для получения последовательности предсказанной изоформы. [9] Изоформа 2 короче из-за укороченного N-конца. Обе изоформы имеют альтернативный сайт полиаденилирования. [8]

регуляция miRNA

miRNA-24 была идентифицирована как микроРНК , которая потенциально может воздействовать на мРНК INAVA. [10] Показан сайт связывания, расположенный в 5'-нетранслируемой области .
Белок
Общие свойства

Изоформа 1, представленная на схеме ниже, содержит домен DUF3338, два участка с низкой сложностью и участок, богатый пролином. Белок богат аргинином и пролином, а также содержит меньшее, чем в среднем, количество аспарагина и гидрофобных аминокислот, в частности фенилаланина и изолейцина. [11] Изоэлектрическая точка составляет 9,58, а молекулярная масса немодифицированного белка составляет 72,9 кдал. [12] Не предполагается, что у белка есть N-концевой сигнальный пептид, но есть предсказанные сигналы ядерной локализации (NLS) и богатый лейцином ядерный экспортный сигнал . [13] [14] [15]
Модификации
INAVA, как предполагается, будет сильно фосфорилирован. [16] [17] Сайты фосфорилирования, предсказанные PROSITE, показаны в таблице ниже. Прогнозы NETPhos проиллюстрированы на диаграмме. Каждая линия указывает на предсказанный сайт фосфорилирования и соединяется с буквой, которая представляет либо серин (S), либо треонин (T), либо тирозин (Y).


Структура
Спиральные спирали, как прогнозируется, охватывают остатки 130-160 и 200-260. [18] Вторичный состав, как прогнозируется, будет включать около 60% случайных спиралей, 30% альфа-спиралей и 10% бета-слоев. [19]
Взаимодействия
Белки, с которыми взаимодействует белок INAVA, недостаточно хорошо охарактеризованы. Данные, полученные в результате анализа текста, свидетельствуют о том, что INAVA может взаимодействовать со следующими белками: DNAJC5G, SLC7A13, PIEZO2, MUC19. [20] Экспериментальные данные, полученные в результате скрининга двух гибридов дрожжей, свидетельствуют о том, что белок INAVA взаимодействует с белком 14-3-3 сигма, который является адаптерным белком. [21]
Гомология
INAVA хорошо сохраняется у позвоночных, как показано в таблице ниже. Последовательности были получены из BLAST [22] и BLAT . [23]
| Последовательность | Род и вид | Общее название | вступление в NCBI | Длина(аа) | Идентичность последовательности | Время с момента расхождения (млн. лет) | |
|---|---|---|---|---|---|---|---|
| * | C1orf106 | Homo sapiens | Человек | NP_060735.3 | 667 | 100% | NA |
| * | C1orf106 | Macaca fascicularis | Макака, поедающая крабов | XP_005540414.1 | 703 | 97% | 29.0 |
| * | LOC289399 | Rattus norvegicus | Норвежская крыса | NP_001178750.1 | 667 | 86% | 92.3 |
| * | Предсказанный гомолог C1orf106 | Одобенус розмарус дивергенс | Морж | XP_004392787.1 | 672 | 85% | 94.2 |
| * | C1orf106-подобный | Loxodonta африканская | Слон | XP_003410255.1 | 663 | 84% | 98.7 |
| * | Предсказанный гомолог C1orf106 | Dasypus novemcinctus | Девятипоясной броненосец | XP_004478752.1 | 676 | 81% | 104.2 |
| * | Предсказанный гомолог C1orf106 | Охотона принцепс | Американская пищуха | XP_004578841.1 | 681 | 78% | 92.3 |
| * | Предсказанный гомолог C1orf106 | Монодельфис домашний | Серый короткохвостый опоссум | XP_001367913.2 | 578 | 76% | 162.2 |
| * | Предсказанный гомолог C1orf106 | Chrysemys picta bellii | Расписная черепаха | XP_005313167.1 | 602 | 56% | 296.0 |
| * | Предсказанный гомолог C1orf106 | Geospiza fortis | Средний земляной вьюрок | XP_005426868.1 | 542 | 50% | 296.0 |
| * | Предсказанный гомолог C1orf106 | Миссисипский аллигатор | Аллигатор | XP_006278041.1 | 547 | 49% | 296.0 |
| * | Предсказанный гомолог C1orf106 | Фицедула белая | Мухоловка-воротничок | XP_005059352.1 | 542 | 49% | 296.0 |
| Предсказанный гомолог C1orf106 | Латимерия лопастная | латимерия западной части Индийского океана | XP_005988436.1 | 613 | 46% | 414.9 | |
| * | Предсказанный гомолог C1orf106 | Леписостеус глазчатый | Пятнистый щука | XP_006628420.1 | 637 | 44% | 400.1 |
| * | Домен FERM, содержащий 4A | Xenopus (Silurana) тропический | Западная когтистая лягушка | XP_002935289.2 | 695 | 43% | 371.2 |
| * | Предсказанный гомолог C1orf106 | Ореохромис нилотический | Нильская тилапия | XP_005478188.1 | 576 | 40% | 400.1 |
| Предсказанный гомолог C1orf106 | Haplochromis burtoni | Астатотиляпия буртони | XP_005914919.1 | 576 | 40% | 400.1 | |
| Предсказанный гомолог C1orf106 | Пундамилия ньеререи | Haplochromis nyererei | XP_005732720.1 | 577 | 40% | 400.1 | |
| * | LOC563192 | Данио рерио | Зебрафиш | NP_001073474.1 | 612 | 37% | 400.1 |
| LOC101161145 | Оризиас латипес | японская рисовая рыба | XP_004069287.1 | 612 | 33% | 400.1 |
Ниже показан график идентичности последовательностей в зависимости от времени с момента расхождения для отмеченных звездочкой записей. Цвета соответствуют степени родства (зеленый = близкородственный, фиолетовый = отдаленнородственный).

Паралоги
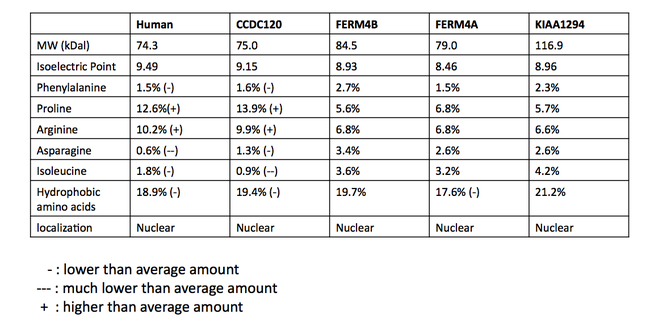
Белки, которые считаются паралогами INAVA, не согласованы между базами данных. Было проведено множественное выравнивание последовательностей (MSA) потенциально паралогичных белков для определения вероятности действительно паралогичных отношений. [24] Последовательности были получены из поиска BLAST у людей с белком C1orf106. MSA предполагает, что белки имеют общий гомологичный домен, DUF3338, который обнаружен у эукариот. Часть множественного выравнивания последовательностей показана ниже. За исключением домена DUF (выделен зеленым), было мало консерватизма. Домен DUF3338 не обладает какими-либо необычными физическими свойствами, однако одним примечательным открытием является то, что каждый из белков в MSA, как предсказано, имеет два сигнала ядерной локализации. Все белки в MSA, как предсказано, локализуются в ядре. [13] Сравнение физических свойств белков также было проведено с использованием SAPS и показано в таблице. [11]

Клиническое значение
В генной области INAVA было выявлено в общей сложности 556 однонуклеотидных полиморфизмов (SNP), 96 из которых связаны с клиническим источником. [25] Ривас и др. [26] выявили четыре SNP, показанные в таблице ниже, которые могут быть связаны с воспалительным заболеванием кишечника и болезнью Крона . Согласно GeneCards, другие ассоциации с заболеваниями могут включать рассеянный склероз и язвенный колит . [27]
| Остаток | Изменять | Примечания |
|---|---|---|
| 333 (rs41313912) | Тирозин ⇒ фенилаланин | Фосфорилированный, умеренная консервация |
| 376 | Аргинин ⇒ цистеин | Умеренная охрана природы |
| 397 | Аргинин ⇒ треонин | Не сохраняется |
| 554 (rs61745433) | Аргинин ⇒ цистеин | Умеренная охрана природы |
Ссылки
- ^ abc GRCh38: Ensembl выпуск 89: ENSG00000163362 – Ensembl , май 2017 г.
- ^ abc GRCm38: Ensembl выпуск 89: ENSMUSG00000041605 – Ensembl , май 2017 г.
- ^ "Human PubMed Reference:". Национальный центр биотехнологической информации, Национальная медицинская библиотека США .
- ^ "Mouse PubMed Reference:". Национальный центр биотехнологической информации, Национальная медицинская библиотека США .
- ^ abcd "NCBI Gene 55765" . Получено 10 февраля 2014 г.
- ^ "Genomatix: MatInspector". Архивировано из оригинала 2 декабря 2021 г. Получено 6 марта 2014 г.
- ^ ab "GEO Profiles" . Получено 6 марта 2014 г.
- ^ ab "Aceview" . Получено 6 марта 2014 г.
- ^ "Softberry" . Получено 20 апреля 2014 г.
- ^ "TargetScanHuman 6.2" . Получено 15 апреля 2014 г.
- ^ ab "Статистический анализ белковых последовательностей" . Получено 20 апреля 2014 г.
- ^ "Compute pI/Mw tool" . Получено 10 апреля 2014 г. .
- ^ ab "PSORTII" . Получено 20 апреля 2014 г.
- ^ "cNLS Mapper". Архивировано из оригинала 22 ноября 2021 г. Получено 20 апреля 2014 г.
- ^ "NetNES" . Получено 20 апреля 2014 г.
- ^ "NETPhos" . Получено 20 апреля 2014 г.
- ^ «Швейцарский институт биоинформатики: PROSITE».
- ^ "ExPASy COILS". Архивировано из оригинала 22 апреля 2014 года . Получено 20 апреля 2014 года .
- ^ "SOPMA" . Получено 27 апреля 2014 г.
- ^ "STRING" . Получено 15 апреля 2014 г.
- ^ "MINT" . Получено 15 апреля 2014 г.
- ^ "BLAST" . Получено 8 марта 2014 г.
- ^ "BLAT" . Получено 8 марта 2014 г.
- ^ "SDSC Biology Workbench: ClustalW" . Получено 12 марта 2014 г.
- ^ "dbSNP" . Получено 22 апреля 2014 г. .
- ^ Ривас МА; и др. (2011). «Глубокое повторное секвенирование локусов GWAS выявляет независимые редкие варианты, связанные с воспалительным заболеванием кишечника». Nature Genetics . 43 (11): 1066– 1073. doi :10.1038/ng.952. PMC 3378381 . PMID 21983784.
- ^ "GeneCards" . Получено 1 мая 2014 г.
Внешние ссылки
- Расположение генома человека C1orf106 и страница с подробностями гена C1orf106 в браузере геномов UCSC .






