База данных генома сахаромицетов
Эта статья может потребовать очистки для соответствия стандартам качества Википедии . Конкретная проблема: Неправильная грамматика. ( Ноябрь 2024 ) |
| Разработчик(и) | Джей Майкл Черри, Гейл Бинкли, Стэйша Энджел, Роб Нэш, Стюарт Миясато, Эдит Вонг, Шуай Вэн |
|---|---|
| Операционная система | Unix , Mac , MS-Windows |
| Тип | Инструмент биоинформатики, база данных модельных организмов |
| Лицензия | Бесплатно |
| Веб-сайт | http://www.yeastgenome.org |
База данных генома Saccharomyces ( SGD ) — это научная база данных молекулярной биологии и генетики дрожжей Saccharomyces cerevisiae , которые обычно называют пекарскими или почковыми дрожжами. [1] Дополнительная информация находится в курируемом репозитории Yeastract . [2]
СахаромицетыБаза данных генома
SGD предоставляет доступ в Интернет к полной геномной последовательности ДНК Saccharomyces cerevisiae , ее генам и их продуктам, фенотипам ее мутантов и литературе, подтверждающей эти данные. В рецензируемом отчете по литературе экспериментальные результаты по функции и взаимодействию генов дрожжей извлекаются с помощью высококачественной ручной обработки и интегрируются в хорошо развитую базу данных. Данные объединяются с качественными высокопроизводительными результатами и размещаются на страницах Locus Summary, которые являются мощным механизмом запросов и богатым браузером генома. Исходя из сложности сбора информации, для интеграции информации и обеспечения продуктивного открытия новых биологических деталей используются многочисленные биоинформатические инструменты. [3] Золотой стандарт для функционального описания почкующихся дрожжей предоставляется SGD. SGD также предоставляет платформу для исследования связанных генов и путей в высших организмах. Объем информации и количество характеристик, предоставляемых SGD, значительно возросли после выпуска геномной последовательности S. cerevisiae . SGD помогает исследователям, предоставляя не только базовую информацию, но и такие инструменты, как поиск сходства последовательностей, которые приводят к подробной информации об особенностях генома и связях между генами. SGD представляет информацию с помощью множества удобных для пользователя, динамически создаваемых графических дисплеев, иллюстрирующих физические, генетические и карты особенностей последовательностей. Все данные в SGD находятся в свободном доступе для исследователей и преподавателей по всему миру через веб-страницы, разработанные для оптимальной простоты использования. [3]
Сбор информации
Biocurator включает обзор опубликованной литературы или наборов данных, что приводит к идентификации и абстрагированию ключевых результатов. Затем результаты включаются в базу данных и используют контролируемые словари, связанные с соответствующими генами или хромосомными областями. По мере сбора большего количества данных биокурирование становится все более важным для биомедицинских исследований.
SGD является источником геномной последовательности для штамма S. cerevisiae S288C и включает каталог генов и хромосомных особенностей.
Одной из важных функций SGD является биокураторство литературы по дрожжам. Биокураторы SGD ищут всю научную литературу, которая имеет отношение к S. cerevisiae , читают статьи и фиксируют свои основные выводы в различных определенных областях базы данных. [3]
Биокураторы SGD стремятся аннотировать каждый ген, идентифицируя функцию(и) из первичной литературы и связывая с терминами, используя структурированное представление знаний в онтологии генов . [4] Кроме того, функции, идентифицированные в ходе высокопроизводительных экспериментов, а также аннотации функций, предсказанные с помощью вычислений, включены в проект аннотаций GO. [5]
Биохимические пути вручную курируются SGD и предоставляются с помощью программного обеспечения Pathway Tools версии 28.0. Набор данных биохимических путей SGD для S. cerevisiae , один из наиболее тщательно курируемых наборов данных среди всех доступных наборов данных Pathway Tools, является золотым стандартом для почкующихся дрожжей; SGD поддерживает постоянные усилия по обновлению и улучшению этих данных. Интерфейс Pathway Tools предоставляет полное описание каждого пути с молекулярными структурами, номерами EC и полным списком ссылок. Обновленный браузер путей предоставляет несколько улучшенных функций, включая загрузку списка генов, обнаруженных в пути, для дальнейшего анализа с помощью других инструментов, доступных в SGD. Браузер путей гиперссылается через раздел «Пути» на странице Locus Summary. [3]
Номенклатура
SGD продолжает поддерживать геномную номенклатуру S. cerevisiae для поддержания стандартов, определенных сообществом, и для обеспечения соблюдения согласованных руководящих принципов при наименовании новых генов или присвоении новых названий ранее идентифицированным генам. Руководящие принципы сообщества гласят, что первое опубликованное название гена становится стандартным названием. Однако перед публикацией название гена может быть зарегистрировано и отображено в SGD, чтобы уведомить сообщество о его предполагаемом использовании. В случае возникновения разногласий или конфликтов названий кураторы SGD связываются с соответствующими исследователями в сообществе и по возможности договариваются о соглашении. Большинство тех, кто работает над рассматриваемым геном, должны согласиться на любое изменение номенклатуры до его внедрения в SGD. Помимо поддержания генетических названий, SGD гарантирует, что названия открытых рамок считывания (ORF), элементов автономно повторяющейся последовательности (ARS), транспортных РНК (tRNA) и других хромосомных признаков также соответствуют согласованным форматам. За последние 2 года было присвоено 154 новых названия генам и обработано 21 изменение названий, инициированное сообществом. [3]
Методы анализа
SGD предоставляет несколько различных инструментов анализа.
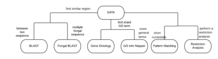
BLAST , Basic Local Alignment Search Tool , — это программа, разработанная для поиска схожих областей между биологическими последовательностями. SGD позволяет пользователям выполнять поиск BLAST в наборах данных последовательностей S. cerevisiae .
Fungal BLAST позволяет проводить поиск среди нескольких последовательностей грибов
Поисковик терминов Gene Ontology (GO) ищет значимые общие термины GO или их родителей и используется для описания запрашиваемых генов, чтобы помочь пользователям узнать, что общего у этих генов.
GO Slim Mapper сопоставляет аннотации группы генов с более общими терминами и/или группирует их в широкие категории.
Сопоставление шаблонов — это ресурс, который позволяет пользователям искать короткие нуклеотидные или пептидные последовательности, состоящие менее чем из 20 остатков, или неоднозначные/вырожденные шаблоны.
Анализ рестрикции позволяет пользователям выполнять анализ рестрикции, вводя имя последовательности или произвольную последовательность ДНК [6]
Ссылки
- ^ Cherry JM; Ball C; Weng S; Juvik G; Schmidt R; Adler C; Dunn B; Dwight S; Riles L; Mortimer RK; Botstein D (май 1997). "Генетические и физические карты Saccharomyces cerevisiae". Nature . 387 (6632 Suppl): 67– 73. doi :10.1038/387s067. PMC 3057085 . PMID 9169866.
- ^ Тейшейра, MC; Монтейро, П; Джайн, П; Тенрейро, С; Фернандес, Арканзас; Мира, НП; Аленкер, М; Фрейтас, АТ; Оливейра, Алабама; Са-Коррейя, I (январь 2006 г.). «База данных YEASTRACT: инструмент для анализа ассоциаций регуляции транскрипции у Saccharomyces cerevisiae». Нуклеиновые кислоты Рез . 34 (Проблема с базой данных). Англия: D446–51. дои : 10.1093/nar/gkj013. ПМЦ 1347376 . ПМИД 16381908.
- ^ abcde Черри, Майкл; Хонг, Эури ; Амундсен, Крейг; Балакришнан, Рама; Бинкли, Гейл; Чан, Эстер; Кристи, Карен; Костанцо, Мария; Дуайт, Селина; Энгель, Стейсия; Фиск, Дианна; Хиршман, Джоди; Хитц, Бенджамин; Карра, Калпана; Кригер, Синтия; Миясато, Стюарт; Нэш, Роб; Парк, Джули; Скржипек, Марек; Симисон, Мэтт; Венг, Шуай; Вонг, Эдит (2011). «База данных геномов Saccharomyces: геномный ресурс почкующихся дрожжей». Nucleic Acids Research . 40 (2012): D700 – D705 . doi : 10.1093/nar/gkr1029. PMC 3245034. PMID 22110037 .
- ^ Дуайт СС, Харрис МА, Долински К и др. (январь 2002 г.). «База данных геномов Saccharomyces (SGD) обеспечивает вторичную аннотацию генов с использованием Gene Ontology (GO)». Nucleic Acids Res . 30 (1): 69– 72. doi :10.1093/nar/30.1.69. PMC 99086. PMID 11752257 .
- ^ Хонг ЕЛ, Балакришнан Р, Донг Кью и др. (январь 2008 г.). «Аннотации генной онтологии в SGD: новые источники данных и методы аннотации». Nucleic Acids Res . 36 (выпуск базы данных): D577–81. doi :10.1093/nar/gkm909. PMC 2238894. PMID 17982175 .
- ^ "База данных генома Saccharomyces". База данных генома Saccharomyces . Стэнфордский университет . Получено 26 апреля 2018 г.
Внешние ссылки
- База данных генома сахаромицетов


