Микроматрица олигонуклеотидов, специфичных для метилирования
Микроматрица олигонуклеотидов метилирования , также известная как микроматрица MSO , была разработана как метод картирования эпигенетических изменений метилирования в ДНК раковых клеток. [1]
Общий процесс начинается с модификации ДНК бисульфитом , в частности, для преобразования неметилированного цитозина в сайтах CpG в урацил, оставляя метилированные цитозины нетронутыми. [1] Модифицированная область интереса ДНК амплифицируется с помощью ПЦР , и в ходе этого процесса урацилы преобразуются в тимин. Ампликоны маркируются флуоресцентным красителем и гибридизуются с олигонуклеотидными зондами , которые фиксируются на предметном стекле. [2] Зонды дифференциально связываются с остатками цитозина и тимина, что в конечном итоге позволяет различать метилированные и неметилированные сайты CpG соответственно. [1]
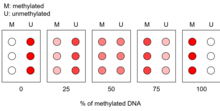
Калибровочная кривая создается и сравнивается с результатами микрочипов амплифицированных образцов ДНК. Это позволяет провести общую количественную оценку доли метилирования, присутствующего в интересующей области. [3]
Этот метод микрочипов был разработан Тимом Хуэй-Мин Хуаном и его лабораторией и был официально опубликован в 2002 году. [1]
Значение для исследований рака
Эта статья нуждается в более надежных медицинских ссылках для проверки или слишком сильно полагается на первичные источники . ( ноябрь 2019 г. ) |
Раковые клетки часто развивают атипичные паттерны метилирования на участках CpG в промоторах генов-супрессоров опухолей . Высокие уровни метилирования на промоторе приводят к снижению регуляции соответствующих генов и характерны для канцерогенеза . Это одно из наиболее последовательных изменений, наблюдаемых в опухолевых клетках на ранней стадии. [1] Микроматрица олигонуклеотидов, специфичных для метилирования, позволяет с высоким разрешением и высокой пропускной способностью обнаруживать многочисленные события метилирования на нескольких промоторах генов. Поэтому этот метод можно использовать для обнаружения аберрантного метилирования в промоторах супрессоров опухолей на ранней стадии, и он использовался при раке желудка и толстой кишки, а также во многих других случаях. [4] [5] Поскольку он позволяет обнаружить наличие атипичных метилирований в раковых клетках, его также можно использовать для выявления основной причины злокачественности, будь то мутации на хромосомах или эпигенетические модификации, а также для выявления того, какие уровни транскрипции генов-супрессоров опухолей затронуты. [2] [6] Интересное применение этого микрочипа включает специфическую классификацию раковых заболеваний, основанную только на паттернах метилирования, например, дифференциацию между классами лейкемии , предполагая, что различные классы рака демонстрируют относительно уникальные паттерны метилирования. [7] Этот метод также был предложен для мониторинга методов лечения рака, которые включают изменение паттернов метилирования в мутантных раковых клетках. [2]
Ссылки
- ^ abcde Gitan RS, Shi H, Chen CM, Yan PS, Huang TH (январь 2002 г.). «Микроматрица олигонуклеотидов, специфичных для метилирования: новый потенциал для высокопроизводительного анализа метилирования». Genome Research . 12 (1): 158– 64. doi :10.1101/gr.202801. PMC 155260 . PMID 11779841.
- ^ abc Shi H, Maier S, Nimmrich I, Yan PS, Caldwell CW, Olek A, Huang TH (январь 2003 г.). «Микрочип на основе олигонуклеотидов для анализа метилирования ДНК: принципы и применение». Журнал клеточной биохимии . 88 (1): 138– 43. doi :10.1002/jcb.10313. PMID 12461783. S2CID 41907903.
- ^ Yan PS, Wei SH, Huang TH (2004). «Микроматрица олигонуклеотидов, специфичных для метилирования». В Tollefsbol TO (ред.). Протоколы эпигенетики . Методы в молекулярной биологии. Т. 287. Humana Press. С. 251– 60. doi :10.1385/1-59259-828-5:251. ISBN 9781592598281. PMID 15273417.
- ^ Hou P, Shen JY, Ji MJ, He NY, Lu ZH (декабрь 2004 г.). «Метод на основе микрочипов для обнаружения изменений метилирования 5'-CpG-островков гена p16(Ink4a) в карциномах желудка». World Journal of Gastroenterology . 10 (24): 3553– 8. doi : 10.3748 /wjg.v10.i24.3553 . PMC 4611991. PMID 15534905.
- ^ Mund C, Beier V, Bewerunge P, Dahms M, Lyko F, Hoheisel JD (апрель 2005 г.). "Анализ на основе массива геномных паттернов метилирования ДНК промотора гена-супрессора опухолей p16INK4A в клеточных линиях карциномы толстой кишки". Nucleic Acids Research . 33 (8): e73. doi :10.1093/nar/gni072. PMC 1087791 . PMID 15860770.
- ^ Yu YP, Paranjpe S, Nelson J, Finkelstein S, Ren B, Kokkinakis D и др. (февраль 2005 г.). «Высокопроизводительный скрининг статуса метилирования генов при раке простаты с использованием массива метилирования олигонуклеотидов». Carcinogenesis . 26 (2): 471– 9. doi : 10.1093/carcin/bgh310 . PMID 15485992.
- ^ Adorján P, Distler J, Lipscher E, Model F, Müller J, Pelet C и др. (март 2002 г.). «Прогнозирование и обнаружение класса опухолей с помощью анализа метилирования ДНК на основе микрочипов». Nucleic Acids Research . 30 (5): 21e–21. doi :10.1093/nar/30.5.e21. PMC 101257. PMID 11861926 .
Внешние ссылки
- Ресурсы, информация и специальные протоколы для анализа метилирования ДНК
- Программное обеспечение для анализа метилирования ДНК


