Метаболом
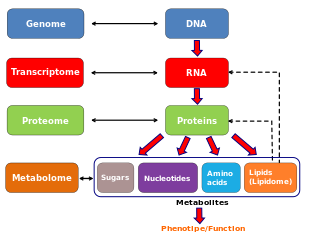
Метаболом относится к полному набору низкомолекулярных химических веществ, обнаруженных в биологическом образце. [1] Биологический образец может быть клеткой , клеточной органеллой , органом , тканью , тканевым экстрактом, биожидкостью или целым организмом . Низкомолекулярные химические вещества, обнаруженные в данном метаболоме, могут включать как эндогенные метаболиты , которые естественным образом вырабатываются организмом (такие как аминокислоты , органические кислоты , нуклеиновые кислоты , жирные кислоты , амины , сахара , витамины , кофакторы , пигменты , антибиотики и т. д.), так и экзогенные химические вещества (такие как лекарства, загрязнители окружающей среды , пищевые добавки , токсины и другие ксенобиотики ), которые естественным образом не вырабатываются организмом. [2] [3]
Другими словами, существует как эндогенный метаболом, так и экзогенный метаболом. Эндогенный метаболом может быть далее подразделен на «первичный» и «вторичный» метаболом (особенно когда речь идет о растительных или микробных метаболомах). Первичный метаболит напрямую участвует в нормальном росте, развитии и воспроизводстве. Вторичный метаболит напрямую не участвует в этих процессах, но обычно имеет важную экологическую функцию. Вторичные метаболиты могут включать пигменты, антибиотики или отходы, полученные из частично метаболизированных ксенобиотиков. Изучение метаболома называется метаболомикой .
Происхождение
Слово метаболом, по-видимому, является слиянием слов «метаболит» и « хромосома ». Оно было создано для того, чтобы подразумевать, что метаболиты косвенно кодируются генами или действуют на гены и продукты генов. Термин «метаболом» был впервые использован в 1998 году [1] [4] и, вероятно, был придуман для соответствия существующим биологическим терминам, относящимся к полному набору генов (геном ) , полному набору белков ( протеом ) и полному набору транскриптов ( транскриптом ). Первая книга по метаболомике была опубликована в 2003 году. [5] Первый журнал, посвященный метаболомике (названный просто «Метаболомика»), был запущен в 2005 году и в настоящее время редактируется профессором Роем Гудакром . Некоторые из наиболее значимых ранних статей по анализу метаболома перечислены в ссылках ниже. [6] [7] [8] [9]
Измерение метаболома
Метаболом отражает взаимодействие между геномом организма и его окружающей средой. В результате метаболом организма может служить отличным зондом его фенотипа (т. е. продукта его генотипа и его окружающей среды). Метаболиты можно измерить (идентифицировать, количественно определить или классифицировать) с помощью ряда различных технологий, включая ЯМР-спектроскопию и масс-спектрометрию . [10] Большинство методов масс-спектрометрии (МС) должны быть связаны с различными формами жидкостной хроматографии (ЖХ), газовой хроматографии (ГХ) или капиллярного электрофореза (КЭ) для облегчения разделения соединений. Каждый метод обычно способен идентифицировать или характеризовать 50-5000 различных метаболитов или «признаков» метаболитов за раз, в зависимости от используемого инструмента или протокола. В настоящее время невозможно проанализировать весь спектр метаболитов одним аналитическим методом.
Спектроскопия ядерного магнитного резонанса (ЯМР) — это метод аналитической химии, который измеряет поглощение радиочастотного излучения определенных ядер, когда молекулы, содержащие эти ядра, помещаются в сильные магнитные поля . Частота (т. е. химический сдвиг ), на которой поглощает данный атом или ядро, в значительной степени зависит от химического окружения (связь, химическая структура ближайших соседей, растворитель) этого атома в данной молекуле. Паттерны поглощения ЯМР создают «резонансные» пики на разных частотах или разных химических сдвигах — этот набор пиков называется спектром ЯМР . Поскольку каждое химическое соединение имеет разную химическую структуру, каждое соединение будет иметь уникальный (или почти уникальный) спектр ЯМР. В результате ЯМР особенно полезен для характеристики, идентификации и количественной оценки малых молекул, таких как метаболиты. Широкое использование ЯМР для «классических» метаболических исследований, наряду с его исключительной способностью обрабатывать сложные смеси метаболитов, вероятно, является причиной того, что ЯМР был одной из первых технологий, широко применяемых для рутинных измерений метаболома. Как аналитический метод ЯМР является неразрушающим, непредвзятым, легко поддается количественной оценке, требует небольшого разделения или не требует его вообще, позволяет идентифицировать новые соединения и не требует химической дериватизации. ЯМР особенно подходит для обнаружения соединений, которые трудно поддаются анализу ЖХ-МС , например, сахара, амины или летучие жидкости, или анализу ГХ-МС , например, большие молекулы (>500 Да) или относительно нереактивные соединения. ЯМР не является очень чувствительным методом с нижним пределом обнаружения около 5 мкМ. Обычно с помощью метаболомных исследований на основе ЯМР можно идентифицировать 50-150 соединений.
Масс-спектрометрия — это аналитический метод, который измеряет отношение массы к заряду молекул. Молекулы или молекулярные фрагменты обычно заряжаются или ионизируются путем распыления их через заряженное поле ( ионизация электрораспылением ), бомбардировки их электронами из горячей нити накала ( ионизация электронов ) или взрыва их лазером, когда они помещаются на специально покрытые пластины (ионизация лазерной десорбцией с помощью матрицы). Затем заряженные молекулы перемещаются в пространстве с помощью электродов или магнитов, и их скорость, скорость кривизны или другие физические характеристики измеряются для определения отношения их массы к заряду. Из этих данных можно определить массу исходной молекулы. Дальнейшая фрагментация молекулы посредством контролируемых столкновений с молекулами газа или с электронами может помочь определить структуру молекул. Очень точные измерения массы также могут использоваться для определения элементарных формул или элементного состава соединений. Большинство форм масс-спектрометрии требуют некоторой формы разделения с использованием жидкостной хроматографии или газовой хроматографии . Этот этап разделения требуется для упрощения результирующих масс-спектров и обеспечения более точной идентификации соединений. Некоторые методы масс-спектрометрии также требуют, чтобы молекулы были дериватизированы или химически модифицированы, чтобы они были более податливы для хроматографического разделения (это особенно актуально для ГХ-МС). Как аналитический метод, МС является очень чувствительным методом, который требует очень малого количества образца (<1 нг материала или <10 мкл биожидкости) и может генерировать сигналы для тысяч метаболитов из одного образца. Инструменты МС также могут быть настроены для очень высокопроизводительного анализа метаболома (сотни-тысячи образцов в день). Количественная оценка метаболитов и характеристика новых структур соединений сложнее для МС, чем для ЯМР. ЖХ-МС особенно подходит для обнаружения гидрофобных молекул ( липидов , жирных кислот) и пептидов, в то время как ГХ-МС лучше всего подходит для обнаружения малых молекул (<500 Да) и высоколетучих соединений ( эфиров , аминов, кетонов , алканов , тиолов ).
В отличие от генома или даже протеома , метаболом является высокодинамичной сущностью, которая может кардинально меняться в течение всего лишь нескольких секунд или минут. В результате растет интерес к измерению метаболитов в течение нескольких периодов времени или в течение коротких временных интервалов с использованием модифицированных версий метаболомики на основе ЯМР или МС.
Базы данных метаболома
Поскольку метаболом организма в значительной степени определяется его геномом, у разных видов будут разные метаболомы. Действительно, тот факт, что метаболом томата отличается от метаболома яблока, является причиной того, почему эти два фрукта имеют такой разный вкус. Более того, разные ткани, разные органы и биожидкости, связанные с этими органами и тканями, также могут иметь отчетливо разные метаболомы. Тот факт, что разные организмы и разные ткани/биожидкости имеют такие разные метаболомы, привел к разработке ряда баз данных метаболомов, специфичных для организмов и биожидкостей. Некоторые из наиболее известных баз данных метаболомов включают Human Metabolome Database или HMDB, [11] Yeast Metabolome Database или YMDB, [12] E. coli Metabolome Database или ECMDB, [13] Arabidopsis metabolome database или AraCyc [14], а также Urine Metabolome Database, [15] Cerebrospinal Fluid (CSF) Metabolome Database [16] и Serum Metabolome Database . [17] Последние три базы данных специфичны для человеческих биологических жидкостей. Существует также ряд очень популярных общих баз данных метаболитов, включая KEGG , [18] MetaboLights, [19] Golm Metabolome Database , [20] MetaCyc , [21] LipidMaps [22] и Metlin . [23] Базы данных метаболомов можно отличить от баз данных метаболитов тем, что базы данных метаболитов содержат слабо аннотированные или синоптические данные о метаболитах из нескольких организмов, в то время как базы данных метаболомов содержат очень подробные и снабженные ссылками данные о химических веществах, путях, спектрах и концентрациях метаболитов для конкретных организмов.
База данных метаболома человека
База данных метаболома человека (HMDB) — это свободно доступная база данных с открытым доступом, содержащая подробные данные о более чем 40 000 метаболитов, которые уже были идентифицированы или, вероятно, будут обнаружены в организме человека. HMDB содержит три вида информации:
- Химическая информация,
- Клиническая информация и
- Биохимическая информация.
Химические данные включают в себя >40 000 структур метаболитов с подробными описаниями, обширные химические классификации, информацию о синтезе и наблюдаемые/расчетные химические свойства. Он также содержит около 10 000 экспериментально измеренных спектров ЯМР , ГХ-МС и ЖХ/МС более 1100 различных метаболитов. Клиническая информация включает в себя данные о >10 000 концентрациях метаболитов-биожидкостей, информацию о концентрации метаболитов для более чем 600 различных заболеваний человека и данные о путях для более чем 200 различных врожденных нарушений метаболизма. Биохимическая информация включает в себя около 6000 последовательностей белков (и ДНК) и более 5000 биохимических реакций, которые связаны с этими записями метаболитов. HMDB поддерживает широкий спектр онлайн-запросов, включая текстовый поиск, поиск химической структуры, поиск сходства последовательностей и поиск спектрального сходства. Это делает его особенно полезным для исследователей метаболомики, которые пытаются идентифицировать или понять метаболиты в клинических метаболомических исследованиях. Первая версия HMDB была выпущена 1 января 2007 года и была составлена учеными из Университета Альберты и Университета Калгари . В то время они сообщили данные о 2500 метаболитах, 1200 лекарственных средствах и 3500 пищевых компонентах. С тех пор эти ученые значительно расширили коллекцию. Версия 3.5 HMDB содержит >16 000 эндогенных метаболитов, >1500 лекарственных средств и >22 000 пищевых компонентов или пищевых метаболитов. [24]
Метаболомы биожидкостей человека
Ученые из Университета Альберты систематически характеризовали специфические метаболомы биожидкостей, включая метаболом сыворотки, [17] метаболом мочи, [15] метаболом спинномозговой жидкости (СМЖ) [16] и метаболом слюны. Эти усилия включали как экспериментальный метаболомный анализ (включая анализы ЯМР, ГХ-МС, ИСП-МС , ЖХ-МС и ВЭЖХ ), так и обширный поиск литературы. Согласно их данным, метаболом сыворотки человека содержит не менее 4200 различных соединений (включая множество липидов), метаболом мочи человека содержит не менее 3000 различных соединений (включая сотни летучих веществ и кишечных микробных метаболитов), метаболом СМЖ человека содержит около 500 различных соединений, в то время как метаболом слюны человека содержит около 400 различных метаболитов, включая множество бактериальных продуктов.
База данных метаболома дрожжей
База данных метаболома дрожжей — это свободно доступная онлайн-база данных, содержащая более 2000 низкомолекулярных метаболитов, обнаруженных или произведенных Saccharomyces cerevisiae ( хлебопекарные дрожжи ). YMDB содержит два вида информации:
- Химическая информация и
- Биохимическая информация.
Химическая информация в YMDB включает 2027 структур метаболитов с подробными описаниями метаболитов, обширные химические классификации, информацию о синтезе и наблюдаемые/вычисленные химические свойства. Он также содержит около 4000 спектров ЯМР, ГХ-МС и ЖХ/МС, полученных из более чем 500 различных метаболитов. Биохимическая информация в YMDB включает >1100 последовательностей белков (и ДНК) и >900 биохимических реакций. YMDB поддерживает широкий спектр запросов, включая текстовый поиск, поиск химической структуры, поиск сходства последовательностей и поиск спектрального сходства. Это делает его особенно полезным для исследователей метаболомики, которые изучают дрожжи как модельный организм или которые ищут оптимизацию производства ферментированных напитков (вино, пиво).
Вторичная электрораспылительная ионизация - масс-спектрометрия высокого разрешения SESI-HRMS - это неинвазивный аналитический метод, который позволяет нам контролировать метаболическую активность дрожжей. SESI-HRMS обнаружил около 300 метаболитов в процессе ферментации дрожжей, это говорит о том, что большое количество метаболитов глюкозы не описано в литературе. [25]
База данных метаболома Escherichia coli
База данных метаболомов E. Coli — это свободно доступная онлайн-база данных, содержащая более 2700 низкомолекулярных метаболитов, обнаруженных или производимых Escherichia coli (штамм E. coli K12, MG1655). ECMDB содержит два вида информации:
- Химическая информация и
- Биохимическая информация.
Химическая информация включает в себя более 2700 структур метаболитов с подробными описаниями метаболитов, обширные химические классификации, информацию о синтезе и наблюдаемые/вычисленные химические свойства. Она также содержит около 5000 спектров ЯМР, ГХ-МС и ЖХ-МС более 600 различных метаболитов. Биохимическая информация включает в себя >1600 последовательностей белков (и ДНК) и >3100 биохимических реакций, которые связаны с этими записями метаболитов. ECMDB поддерживает множество различных типов онлайн-запросов, включая текстовый поиск, поиск химической структуры, поиск сходства последовательностей и поиск спектрального сходства. Это делает ее особенно полезной для исследователей метаболомики, которые изучают E. coli как модельный организм.
Вторичная электрораспылительная ионизация (SESI-MS) позволяет различать одиннадцать штаммов E. Coli благодаря профилированию летучих органических соединений. [26]
Атлас метаболома стареющего мозга мыши
В 2021 году первый атлас метаболома мозга мыши и животного (млекопитающего) на разных этапах жизни был опубликован в сети. Данные различаются по областям мозга, а метаболические изменения могут быть «сопоставлены с существующими атласами генов и белков мозга». [27] [28]
Кишечный метаболом
Микробиота кишечника человека вносит вклад в этиологию колоректального рака посредством своего метаболома. [29] В частности, преобразование первичных желчных кислот во вторичные желчные кислоты в результате бактериального метаболизма в толстой кишке способствует канцерогенезу . [29]
Смотрите также
Ссылки
- ^ ab Oliver SG, Winson MK, Kell DB, Baganz F (сентябрь 1998 г.). «Систематический функциональный анализ генома дрожжей». Trends in Biotechnology . 16 (9): 373– 8. CiteSeerX 10.1.1.33.5221 . doi :10.1016/S0167-7799(98)01214-1. PMID 9744112.
- ^ Wishart DS (сентябрь 2007 г.). «Текущий прогресс в вычислительной метаболомике». Briefings in Bioinformatics . 8 (5): 279–93 . doi : 10.1093/bib/bbm030 . PMID 17626065.
- ^ Nordström A, O'Maille G, Qin C, Siuzdak G (май 2006 г.). «Нелинейное выравнивание данных для метаболомики на основе UPLC-MS и HPLC-MS: количественный анализ эндогенных и экзогенных метаболитов в сыворотке человека». Аналитическая химия . 78 (10): 3289– 95. doi :10.1021/ac060245f. PMC 3705959 . PMID 16689529.
- ^ Tweeddale H, Notley-McRobb L, Ferenci T (октябрь 1998 г.). «Влияние медленного роста на метаболизм Escherichia coli, выявленное с помощью анализа глобального метаболитного пула («метаболома»)». Журнал бактериологии . 180 (19): 5109– 16. doi :10.1128/JB.180.19.5109-5116.1998. PMC 107546. PMID 9748443.
- ^ Harrigan GG, Goodacre R, ред. (2003). Метаболическое профилирование: его роль в обнаружении биомаркеров и анализе функций генов . Бостон: Kluwer Academic Publishers. ISBN 978-1-4020-7370-0.
- ^ Fiehn O, Kloska S, Altmann T (февраль 2001 г.). «Комплексные исследования биологии растений с использованием многопараллельных методов». Current Opinion in Biotechnology . 12 (1): 82– 6. doi :10.1016/S0958-1669(00)00165-8. PMID 11167078.
- ^ Fiehn O (2001). «Объединение геномики, анализа метаболома и биохимического моделирования для понимания метаболических сетей». Сравнительная и функциональная геномика . 2 (3): 155–68 . doi :10.1002/cfg.82. PMC 2447208. PMID 18628911 .
- ^ Weckwerth W (2003). «Метаболомика в системной биологии». Annual Review of Plant Biology . 54 : 669–89 . doi :10.1146/annurev.arplant.54.031902.135014. PMID 14503007. S2CID 1197884.
- ^ Goodacre R, Vaidyanathan S, Dunn WB, Harrigan GG, Kell DB (май 2004 г.). «Метаболомика в цифрах: получение и понимание глобальных данных о метаболитах». Trends in Biotechnology . 22 (5): 245–52 . doi :10.1016/j.tibtech.2004.03.007. PMID 15109811.
- ^ Lu W, Su X, Klein MS, Lewis IA, Fiehn O, Rabinowitz JD (июнь 2017 г.). «Измерение метаболитов: подводные камни, которых следует избегать, и методы, которым следует следовать». Annual Review of Biochemistry . 86 (1): 277– 304. doi :10.1146/annurev-biochem-061516-044952. PMC 5734093. PMID 28654323 .
- ^ Wishart DS, Tzur D, Knox C, Eisner R, Guo AC, Young N и др. (январь 2007 г.). "HMDB: база данных метаболома человека". Nucleic Acids Research . 35 (выпуск базы данных): D521-6. doi :10.1093/nar/gkl923. PMC 1899095. PMID 17202168 .
- ^ Jewison T, Knox C, Neveu V, Djoumbou Y, Guo AC, Lee J, et al. (Январь 2012). "YMDB: база данных метаболома дрожжей". Nucleic Acids Research . 40 (выпуск базы данных): D815-20. doi :10.1093/nar/gkr916. PMC 3245085. PMID 22064855 .
- ^ Guo AC, Jewison T, Wilson M, Liu Y, Knox C, Djoumbou Y и др. (январь 2013 г.). "ECMDB: база данных метаболома E. coli". Nucleic Acids Research . 41 (выпуск базы данных): D625-30. doi :10.1093/nar/gks992. PMC 3531117. PMID 23109553 .
- ^ Mueller LA, Zhang P, Rhee SY (июнь 2003 г.). "AraCyc: база данных биохимических путей для Arabidopsis". Plant Physiology . 132 (2): 453– 60. doi :10.1104/pp.102.017236. PMC 166988. PMID 12805578 .
- ^ ab Bouatra S, Aziat F, Mandal R, Guo AC, Wilson MR, Knox C и др. (сентябрь 2013 г.). "Метаболом мочи человека". PLOS ONE . 8 (9): e73076. Bibcode :2013PLoSO...873076B. doi : 10.1371/journal.pone.0073076 . PMC 3762851 . PMID 24023812.
- ^ ab Mandal R, Guo AC, Chaudhary KK, Liu P, Yallou FS, Dong E и др. (апрель 2012 г.). «Мультиплатформенная характеристика метаболома спинномозговой жидкости человека: комплексное и количественное обновление». Genome Medicine . 4 (4): 38. doi : 10.1186/gm337 . PMC 3446266 . PMID 22546835.
- ^ ab Psychogios N, Hau DD, Peng J, Guo AC, Mandal R, Bouatra S, et al. (февраль 2011 г.). "Метаболом сыворотки человека". PLOS ONE . 6 (2): e16957. Bibcode :2011PLoSO...616957P. doi : 10.1371/journal.pone.0016957 . PMC 3040193 . PMID 21359215.
- ^ Канехиса М., Гото С. (январь 2000 г.). «KEGG: киотская энциклопедия генов и геномов». Nucleic Acids Research . 28 (1): 27– 30. doi :10.1093/nar / 28.1.27. PMC 102409. PMID 10592173.
- ^ Haug K, Salek RM, Conesa P, Hastings J, de Matos P, Rijnbeek M и др. (январь 2013 г.). "MetaboLights — общедоступный репозиторий общего назначения для исследований метаболомики и связанных с ними метаданных". Nucleic Acids Research . 41 (выпуск базы данных): D781-6. doi :10.1093/nar/gks1004. PMC 3531110. PMID 23109552.
- ^ Копка Дж., Шауэр Н., Крюгер С., Биркемейер К., Усадель Б., Бергмюллер Э. и др. (апрель 2005 г.). «GMD@CSB.DB: база данных метаболома Гольма». Биоинформатика . 21 (8): 1635– 8. doi : 10.1093/bioinformatics/bti236 . hdl : 20.500.11850/33179 . PMID 15613389.
- ^ Caspi R, Altman T, Dale JM, Dreher K, Fulcher CA, Gilham F и др. (январь 2010 г.). «База данных метаболических путей и ферментов MetaCyc и коллекция баз данных путей/геномов BioCyc». Nucleic Acids Research . 38 (выпуск базы данных): D473-9. doi :10.1093/nar/gkp875. PMC 2808959. PMID 19850718 .
- ^ Fahy E, Sud M, Cotter D, Subramaniam S (июль 2007 г.). "LIPID MAPS онлайн-инструменты для исследования липидов". Nucleic Acids Research . 35 (выпуск веб-сервера): W606-12. doi :10.1093/nar/gkm324. PMC 1933166. PMID 17584797 .
- ^ Smith CA, O'Maille G, Want EJ, Qin C, Trauger SA, Brandon TR и др. (декабрь 2005 г.). «METLIN: база данных масс-спектров метаболитов». Therapeutic Drug Monitoring . 27 (6): 747– 51. doi :10.1097/01.ftd.0000179845.53213.39. PMID 16404815. S2CID 14774455.
- ^ Wishart DS, Jewison T, Guo AC, Wilson M, Knox C, Liu Y и др. (январь 2013 г.). "HMDB 3.0 — база данных метаболома человека в 2013 г.". Nucleic Acids Research . 41 (выпуск базы данных): D801-7. doi :10.1093/nar/gks1065. PMC 3531200. PMID 23161693 .
- ^ Tejero Rioseras A, Garcia Gomez D, Ebert BE, Blank LM, Ibáñez AJ, Sinues PM (октябрь 2017 г.). "Комплексный анализ дрожжевого летучего вещества в реальном времени". Scientific Reports . 7 (1): 14236. Bibcode :2017NatSR...714236T. doi :10.1038/s41598-017-14554-y. PMC 5660155 . PMID 29079837.
- ^ Zhu J, Hill JE (июнь 2013 г.). «Обнаружение Escherichia coli с помощью профилирования ЛОС с использованием вторичной электрораспылительной ионизации-масс-спектрометрии (SESI-MS)». Пищевая микробиология . 34 (2): 412– 7. doi :10.1016/j.fm.2012.12.008. PMC 4425455. PMID 23541210 .
- ^ "Карта метаболизма мозга мышей при старении". Калифорнийский университет в Дэвисе . Получено 15 ноября 2021 г.
- ^ Ding J, Ji J, Rabow Z, Shen T, Folz J, Brydges CR и др. (октябрь 2021 г.). «Атлас метаболома мозга стареющей мыши». Nature Communications . 12 (1): 6021. Bibcode : 2021NatCo..12.6021D. doi : 10.1038/s41467-021-26310-y . PMC 8519999. PMID 34654818 .
- ^ ab Louis P, Hold GL, Flint HJ (октябрь 2014 г.). «Микробиота кишечника, бактериальные метаболиты и колоректальный рак». Nature Reviews. Microbiology . 12 (10): 661–72 . doi :10.1038/nrmicro3344. PMID 25198138.
