ФАМ76А
| ФАМ76А | |||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Идентификаторы | |||||||||||||||||||||||||||||||||||||||||||||||||||
| Псевдонимы | FAM76A , семейство с последовательностью сходства 76 член A | ||||||||||||||||||||||||||||||||||||||||||||||||||
| Внешние идентификаторы | MGI : 2385211; HomoloGene : 27071; GeneCards : FAM76A; OMA :FAM76A - ортологи | ||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Викиданные | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
FAM76A — это белок, который у Homo sapiens кодируется геном FAM76A . [5] Известные структурные характеристики FAM76A включают домен спиральной спирали из 83 аминокислот , а также смещение состава полисерина из четырех аминокислот. [6] FAM76A сохраняется у большинства хордовых , но не встречается у других представителей deuterostrome phlya, таких как echinodermata , hemichordata или xenacoelomorpha — что позволяет предположить, что FAM76A возник в эволюционной линии через некоторое время после хордовых. Кроме того, FAM76A не встречается у грибов , растений , архей или бактерий . [7] Предполагается, что FAM76A локализуется в ядре и может играть роль в регуляции транскрипции . [8]
Ген
Расположение

FAM76A расположен на (+) цепи короткого плеча хромосомы 1 (1p35.3), с геномной последовательностью, начинающейся с 27725979 и заканчивающейся на 27762915. Кодирующая область состоит из 3462 пар оснований и транслируется в 341 аминокислоту. [5] [10]
Джин соседство
Гены, которые фланкируют FAM76A с теломерной стороны, включают IFI6, CHMP1AP1 и RPEP3, в то время как гены, которые фланкируют FAM76A с центромерной стороны, включают STX12 , PPP1R8 и L0C105376894. [5]
Распространенные псевдонимы
У Caenorhabditis elegans FAM76A обозначается как K04F10.7. [11] За исключением этого, FAM76A не имеет никаких значимых альтернативных названий.
мРНК
У Homo sapiens ген FAM76A производит 9 различных мРНК, 7 из которых альтернативно сплайсированы, а 2 — не сплайсированы. Из альтернативно сплайсированных мРНК изоформа 1 является самым длинным вариантом гена и является предметом этой статьи. [5]
Белок
Общие свойства
Молекулярный вес FAM76A составляет 38,4 кДа , что позволяет этому белку диффундировать через ядерные поры . [12] Изоэлектрическая точка составляет 9,28. FAM76A не имеет каких-либо значительных положительных, отрицательных или смешанных зарядовых кластеров. Кроме того, FAM76A не имеет никаких предсказанных гидрофобных или трансмембранных сегментов, что позволяет предположить, что этот белок не обнаружен внутри клеточной мембраны. [13]
Состав
Аминокислотный состав белка FAM76A показал частоты аминокислот в пределах 1,5% от таковых нормальных человеческих белков для всех, кроме цистеина, валина и лизина. Цистеин и лизин имеют более высокие частоты по сравнению с нормальным белком Homo sapiens , в то время как валин имеет более низкую частоту по сравнению с нормальным белком Homo sapiens . Эти же различия в частотах аминокислот наблюдаются в ортологах FAM76A, таких как Gallus gallus ( идентичность последовательности H. sapiens 84%), Serinus canaria ( идентичность последовательности H. sapiens 77%) и Crassostrea gigas ( идентичность последовательности H. sapiens 57%).
Домены и мотивы

Поиск консервативных доменов NCBI выявил неохарактеризованный консервативный белок (YqiK), содержащий домен Band7/PHB/SPFH, функция которого неизвестна и сохраняется у различных видов, от людей до бактерий. [10] У Homo sapiens домен Band7/PHB/SPFH охватывает аминокислоты 252-326. Молекулярная масса этого домена составляет 8,9 кДа, а его изоэлектрическая точка составляет 9,23. Домен Band7/PHB/SPFH не имеет какого-либо состава частот аминокислот, который отличался бы от обычного белка Homo sapiens . [13] Этот домен еще не отнесен ни к одному суперсемейству доменов.
Вторичная структура

FAM76A, как предсказывают, имеет только альфа-спирали. Всего предсказано 17 альфа-спиралей, самая длинная из которых содержит домен Band7/PHB/SPFH. [14] Из этого следует, что только 8 альфа-спиралей расположены в консервативных областях FAM76A (см. концептуальный перевод).
Третичная/четвертичная структура
FAM76A содержит домен спиральной спирали, который расположен в домене Band7/PHB/SPFH. Никаких существенных участков связывания лиганда или активных участков не было предсказано с помощью I-TASSER. [15] Нет никаких доказательств, позволяющих предположить, что FAM76A взаимодействует с другими белками с образованием четвертичной структуры .
Субклеточная локализация
Инструмент прогнозирования субклеточной локализации белка PSORT II предсказывает, что FAM76A будет расположен в ядре. Это предсказание наблюдается у ортологов, таких как Gallus gallus и Callorhinchus milii . [16] Дополнительные доказательства локализации FAM76A в ядре предоставляются наличием сигнала ядерной локализации. [8]
Выражение
Согласно NCBI Geo Profile, FAM76A экспрессируется в паращитовидной железе , лимфатических узлах , пищеводе и костном мозге Homo sapiens . Стадии развития, на которых обнаруживается экспрессия FAM76A, включают эмбриональное тело, плод и взрослую особь. [17]
Атлас мозга
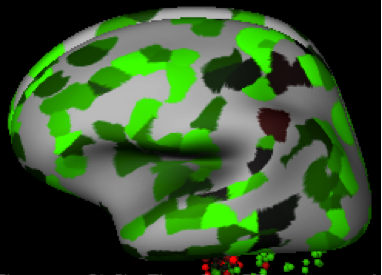
Ниже представлены прогнозы атласа человеческого мозга Аллена для экспрессии FAM76A. FAM76A, по-видимому, имеет более высокую экспрессию в коре головного мозга и более низкую экспрессию в таких частях мозга рептилий , как покрышка моста (см. таблицу экспрессии для получения более подробной информации). [18]
- Различные виды экспрессии FAM76A в мозге Homo sapiens: зеленый цвет обозначает высокую экспрессию, а красный — низкую экспрессию.
- Вид спереди
- Каудальный вид
- Сагиттальный вид
| Область мозга | Функция | Уровень экспрессии FAM76A |
|---|---|---|
| Лобная доля | Планирование, организация, решение проблем, избирательное внимание, личность и высшие когнитивные функции | Высокий |
| Затылочная доля | Визуальная обработка | Высокий |
| Височная доля | Обработка слуховой информации | Высокий |
| Теменная доля | Ощущение, восприятие и интеграция сенсорной информации | Высокий |
| Мозжечок | Координация произвольных движений | Высокий |
| Формирование гиппокампа | Память/пространственное кодирование | Низкий |
| Покрышка моста | Сенсорная и двигательная функция, контроль стадий сна и возбуждение | Низкий |
| Ядро клиновидного продолговатого мозга | Получайте тонкую тактильную и проприоцептивную информацию от верхней части тела | Низкий |
Экспериментальные данные
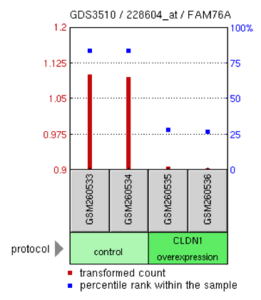
Ниже приведены некоторые данные из трех экспериментов с участием FAM76A. В одном эксперименте повышенная экспрессия CLDN1 в клетках аденокарциномы легких снизила экспрессию FAM76A. [19] В другом эксперименте было показано, что нечувствительные к андрогенам клетки рака простаты имеют пониженную экспрессию FAM76A по сравнению с чувствительными к андрогенам клетками. [20] Другой эксперимент продемонстрировал, что клетки ооцитов метафазы II имеют большую экспрессию FAM76A по сравнению с контрольными клетками. [21]
- Избранные геоданные NCBI
- Уровни FAM76A в клеточных линиях аденокарциномы легких H. sapiens сравнивались между культурами, в которых наблюдалась сверхэкспрессия клаудина-1 (CLDN1), и контрольными клетками.
- Экспрессия FAM76A в линиях клеток рака простаты Homo sapiens, чувствительных и нечувствительных к андрогенам
- Экспрессия FAM76A между ооцитами метафазы II H. sapiens и контрольными линиями клеток (состоящими из смеси скелетных мышц, почек, легких, толстой кишки, печени, селезенки, молочной железы, мозга, сердца и желудка)
Регулирование экспрессии
Посттрансляционные модификации
FAM76A, как прогнозируется, подвергается различным посттрансляционным модификациям. Посттрансляционные модификации, обнаруженные в консервативных областях, включают 7 сайтов фосфорилирования, 2 сайта сумоилирования и 1 сигнал ядерной локализации. [22] Эти модификации указывают на то, что FAM76A локализован в ядре. Обратитесь к концептуальному переводу для визуального представления вышеупомянутых модификаций.
- Концептуальный перевод для FAM76A
- Концептуальный ключ перевода
Промоутер
Программа ElDorado Genomatrix предсказывает промотор для FAM76A, который называется GXP_71042 и состоит из 679 пар оснований. Он расположен на хромосоме 1, начиная с 27725479 и заканчивая 27726157. GXP_71042 перекрывается с началом кодирующей последовательности FAM76A. [23] Существует несколько факторов транскрипции, которые связываются с этим промотором. Многие из факторов транскрипции, которые связываются с промоторной областью FAM76A, имеют функцию, связанную с клетками крови , иммунной системой и лейкоцитами — возможно, предполагая, что FAM76A участвует в иммунной функции. Также, по-видимому, наиболее распространенные семейства матриц включают цинковые пальцы C2H2 и миелоидные цинковые пальцы, предполагая, что эти семейства матриц могут быть активно вовлечены в транскрипцию FAM76A.
РНК-связывающие белки
Распространенные РНК-связывающие белки в 3'-нетранслируемой области FAM76A включают PABPC1 , ELAVL1 и PUM2 — каждый из которых имеет прогнозируемую частоту связывания 32, 18 и 16 раз соответственно. [24]
Взаимодействующие белки
Было обнаружено, что FAM76A имеет физическое взаимодействие с ELAVL1 . Взаимодействие было обнаружено методом иммунопреципитации Абдельмохсеном и др., 2009. [25] ELAVL1 участвует в регуляции экспрессии генов.
Гомология/эволюция
Паралоги
FAM76B является паралогом FAM76A. По оценкам, FAM76A и FAM76B разошлись друг от друга около 17,5 млн лет назад. [5] Структурные сходства, которые сохраняются между FAM76A/B, включают домен спиральной спирали, а также смещение состава полисерина. [10] FAM76A и FAM76B оба демонстрируют высокую экспрессию в таких тканях, как лимфатический узел, цельная кровь, яички, яичник, мозг, почки, печень и легкие. [26] FAM76B имеет около 62% идентичности последовательности с FAM76A. [10]
Ортологи
| Род и вид | Общее название | Дата расхождения (MYA) | Идентичность аминокислотной последовательности (%) |
| Homo sapiens | люди | 0 | 100 |
| Macaca fascicularis | макака-крабоед | 29.1 | 95 |
| Tarsius syrichta | Филиппинский долгопят | 67.6 | 85 |
| Dipodomys ordii | Кенгуровая крыса Орда | 90,9 | 85 |
| Нанноспалакс галили | слепой землекоп | 90,9 | 88 |
| Галлус галлус | красная джунглевая курица | 320,5 | 84 |
| Ниппония ниппон | хохлатый ибис | 320,5 | 83 |
| Белая цапля гарцетта | маленькая цапля | 320,5 | 75 |
| Анолис каролинский | каролинский анолис | 320,5 | 73 |
| Оризиас латипес | японская рисовая рыба | 429,6 | 59 |
| Callorhinchus milii | Австралийская акула-призрак | 482.9 | 64 |
| Крассострея гигантская | тихоокеанская устрица | 847 | 57 |
| Cryptosporidium parvum Айова II | Н/Д | 1724.7 | 27 |
| Криптоспоридии хоминис | Н/Д | 1724.7 | 26 |
Здесь показана таблица избранного числа ортологов для Homo sapiens FAM76A. Таблица включает близкородственные, промежуточные и отдаленнородственные ортологи. Показано, что млекопитающие имеют большее сходство, в то время как водные позвоночные, такие как лучепёрые рыбы / хрящевые рыбы, имеют меньшее сходство. Ортологи белка Homo sapiens FAM76A перечислены выше в порядке убывания даты расхождения, а затем по идентичности последовательностей.
Эволюция
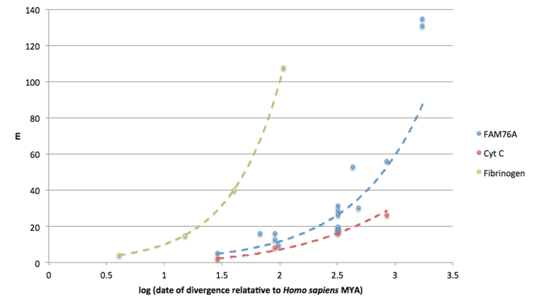
FAM76A, по-видимому, имеет умеренную скорость мутаций по сравнению с фибриногеном (быстро мутирующим) и цитохромом c (медленно мутирующим). [7] [27] Это говорит о том, что FAM76A был, по крайней мере, в некоторой степени устойчив к мутациям в ходе эволюции.
Клиническое значение
Ассоциация заболеваний
Экспрессия FAM76A наиболее высока в опухолях надпочечников , опухолях пищевода и опухолях мягких тканей/мышечной ткани. [5] [28] Увеличение/утрата числа копий FAM76A — вместе с соседними генами — приводит к появлению вредных фенотипов. В одном отчете о случае у пациента с увеличением числа копий от 1p36.11 до 34.2 наблюдались задержки в развитии. [29] Другой пациент с увеличением числа копий от 1p36.1 до 35 показал схожие задержки. [30] В другом отчете о случае у пациента с потерей числа копий 1p35.3, точного местоположения FAM76A, развилась макроцефалия . [30]
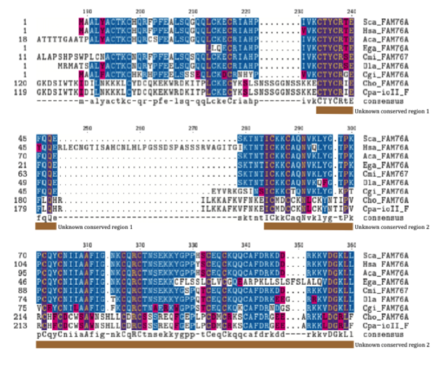
Множественное выравнивание последовательностей (MSA)
MSA, показанная ниже и созданная с помощью Biology Workbench CLUSTALW , упорядочивает ортологов по первой букве рода, а затем по первым двум буквам вида. [13] Существует 3 домена, которые высококонсервативны среди ортологов. Два из этих доменов имеют неизвестную функцию, в то время как третий домен является доменом спиральной спирали. Консервация этих регионов была прослежена до Cryptosporidium parvum Iowa II , который отделился от Homo sapiens 1724,7 млн лет назад. Консервативный регион 1 содержит в основном полярные аминокислоты; консервативный регион 2 содержит как полярные, так и неполярные аминокислоты; а домен спиральной спирали содержит в основном полярные аминокислоты.
- Выравнивание множественных последовательностей дальних ортологов FAM76A
- Фиолетовый=Похожая аминокислотная химия
Синий=Та же аминокислота
Ссылки
- ^ abc GRCh38: Ensembl выпуск 89: ENSG00000009780 – Ensembl , май 2017 г.
- ^ abc GRCm38: Ensembl выпуск 89: ENSMUSG00000028878 – Ensembl , май 2017 г.
- ^ "Human PubMed Reference:". Национальный центр биотехнологической информации, Национальная медицинская библиотека США .
- ^ "Mouse PubMed Reference:". Национальный центр биотехнологической информации, Национальная медицинская библиотека США .
- ^ abcdef "FAM76A family with sequence similarity 76 member A [Homo sapiens (human)] - Gene - NCBI". www.ncbi.nlm.nih.gov . Получено 2016-02-08 .
- ^ "FAM76A Uniprot Entry".
- ^ ab "NCBI BLAST".
- ^ ab "NLS Mapper". Архивировано из оригинала 2021-11-22.
- ^ «Запись гена NCBI FAM76A».
- ^ abcd "белок FAM76A изоформа 1 [Homo sapiens] - Белок - NCBI". www.ncbi.nlm.nih.gov . Получено 2016-02-08 .
- ^ "Профиль Wormbase K04F10.7".
- ^ Махаджан Р., Дельфин К., Гуан Т., Джерейс Л., Мельхиор Ф. (январь 1997 г.). «Небольшой полипептид, связанный с убиквитином, участвующий в нацеливании RanGAP1 на комплексный белок ядерной поры RanBP2». Cell . 88 (1): 97– 107. doi : 10.1016/S0092-8674(00)81862-0 . PMID 9019411. S2CID 17819277.
- ^ abc "SDSC Biology Workbench". workbench.sdsc.edu . Получено 28.02.2016 .
- ^ "Файр2".[ постоянная мертвая ссылка ]
- ^ «Прогнозы структуры и функций белка I-TASSER» .
- ^ «Прогноз PSORT II».
- ^ «Геопрофиль NCBI».
- ^ «Атлас мозга Аллена FAM76A».
- ^ Chao YC, Pan SH, Yang SC, Yu SL, Che TF, Lin CW, Tsai MS, Chang GC, Wu CH, Wu YY, Lee YC, Hong TM, Yang PC (январь 2009 г.). «Клаудин-1 является супрессором метастазов и коррелирует с клиническим исходом при аденокарциноме легких». Американский журнал респираторной и интенсивной терапии . 179 (2): 123– 33. doi :10.1164/rccm.200803-456OC. PMID 18787218.
- ^ Zhao H, Kim Y, Wang P, Lapointe J, Tibshirani R, Pollack JR, Brooks JD (май 2005 г.). «Геномная характеристика вариаций экспрессии генов и изменений числа копий ДНК в клеточных линиях рака простаты». The Prostate . 63 (2): 187–97 . doi :10.1002/pros.20158. PMID 15486987. S2CID 31990854.
- ^ Kocabas AM, Crosby J, Ross PJ, Otu HH, Beyhan Z, Can H, Tam WL, Rosa GJ, Halgren RG, Lim B, Fernandez E, Cibelli JB (сентябрь 2006 г.). «Транскриптом человеческих ооцитов». Труды Национальной академии наук Соединенных Штатов Америки . 103 (38): 14027– 32. Bibcode : 2006PNAS..10314027K. doi : 10.1073/pnas.0603227103 . PMC 1599906. PMID 16968779 .
- ^ "Посттрансляционные модификации ExPasy".
- ^ «Геноматрица Эльдорадо».[ постоянная мертвая ссылка ]
- ^ «RBPDP: База данных специфичностей РНК-связывающих белков».
- ^ Абдельмохсен К., Шрикантан С., Ян Х, Лал А., Ким Х.Х., Кувано Ю., Гальбан С., Беккер К.Г., Камара Д., де Кабо Р., Гороспе М. (май 2009 г.). «Убиквитин-опосредованный протеолиз HuR путем теплового шока». Журнал ЭМБО . 28 (9): 1271–82 . doi :10.1038/emboj.2009.67. ПМК 2683047 . ПМИД 19322201.
- ^ "NCBI AceView".
- ^ «Древо времени».
- ^ «Карты генов FAM76A».
- ^ Miller DT, Adam MP, Aradhya S, Biesecker LG, Brothman AR, Carter NP, Church DM, Crolla JA, Eichler EE, Epstein CJ, Faucett WA, Feuk L, Friedman JM, Hamosh A, Jackson L, Kaminsky EB, Kok K, Krantz ID, Kuhn RM, Lee C, Ostell JM, Rosenberg C, Scherer SW, Spinner NB, Stavropoulos DJ, Tepperberg JH, Thorland EC, Vermeesch JR, Waggoner DJ, Watson MS, Martin CL, Ledbetter DH (май 2010 г.). «Консенсусное заявление: хромосомный микроматричный анализ является клиническим диагностическим тестом первого уровня для лиц с нарушениями развития или врожденными аномалиями». American Journal of Human Genetics . 86 (5): 749– 64. doi : 10.1016/j.ajhg.2010.04.006. PMC 2869000. PMID 20466091 .
- ^ ab Kaminsky EB, Kaul V, Paschall J, Church DM, Bunke B, Kunig D, Moreno-De-Luca D, Moreno-De-Luca A, Mulle JG, Warren ST, Richard G, Compton JG, Fuller AE, Gliem TJ, Huang S, Collinson MN, Beal SJ, Ackley T, Pickering DL, Golden DM, Aston E, Whitby H, Shetty S, Rossi MR, Rudd MK, South ST, Brothman AR, Sanger WG, Iyer RK, Crolla JA, Thorland EC, Aradhya S, Ledbetter DH, Martin CL (сентябрь 2011 г.). «Доказательный подход к установлению функционального и клинического значения вариантов числа копий при нарушениях интеллектуального и развития». Генетика в медицине . 13 (9): 777– 84. doi :10.1097/GIM.0b013e31822c79f9. PMC 3661946. PMID 21844811 .
Дальнейшее чтение
- Абдельмохсен К., Шрикантан С., Ян Х., Лал А., Ким Х.Х., Кувано Ю., Гальбан С., Беккер К.Г., Камара Д., де Кабо Р., Гороспе М. (май 2009 г.). «Убиквитин-опосредованный протеолиз HuR путем теплового шока». Журнал ЭМБО . 28 (9): 1271–82 . doi :10.1038/emboj.2009.67. ПМК 2683047 . ПМИД 19322201.
- Kocabas AM, Crosby J, Ross PJ, Otu HH, Beyhan Z, Can H, Tam WL, Rosa GJ, Halgren RG, Lim B, Fernandez E, Cibelli JB (сентябрь 2006 г.). «Транскриптом человеческих ооцитов». Труды Национальной академии наук Соединенных Штатов Америки . 103 (38): 14027– 32. Bibcode : 2006PNAS..10314027K . doi : 10.1073/pnas.0603227103 . PMC 1599906. PMID 16968779.